导读
单细胞ATAC-seq(scATAC-seq)是一种强大的技术,用于以单细胞分辨率探究表观基因组景观。然而,从scATAC-seq数据推断细胞的确切发育轨迹是具有挑战性的。
虽然存在用于确定细胞进化轨迹的工具,如在单细胞RNA测序(scRNA-seq)数据集中,干细胞预测和代谢标记可用于确定多种表型上的细胞进化轨迹,但scATAC-seq则没有类似的方法。
谱系推断方法在表型具有流动性时会受到限制,例如在去分化或癌变过程中中。另一方面,基于突变的谱系追踪方法,例如使用线粒体单核苷酸多态性(SNPs),可以跟踪细胞在分裂过程中的谱系,具有高度的准确性,但其时间分辨率受到低自然突变率的限制。
有关染色体可及性与DNA甲基化(DNAm)之间内在联系的概念表明,年龄依赖的DNAm可能是导致时钟样差异甲基化位点(ClockDML)的染色体可及性变化。如果是这样,那么来自scATAC-seq数据的单细胞的衍生有丝分裂年龄将成为描绘发育轨迹的强大工具。
理论上,有丝分裂年龄是一个“时间管理”跟踪器:祖先细胞的有丝分裂年龄低于其后代,较早产生的细胞的有丝分裂年龄应低于较晚产生的细胞。如果存在这样的细胞年龄度量,将为复杂生物体中的细胞出生序列提供精确的时间参考,以帮助描绘发育轨迹。
2024年5月9日,Euler科技Yi Zhang联合武汉大学中南医院泌尿外科王行环团队在Nature Biotechnology上发表了一篇题为“Tracking single-cell evolution using clock-like chromatin accessibility loci”的论文,文章们利用上述思路,提出了一个新的计算框架,名为EpiTrace,它可以通过计算单个细胞中特定位点的打开比例来推断细胞的有丝分裂年龄,从而描绘其发育轨迹。

文章索引
【标题】Tracking single-cell evolution using clock-like chromatin accessibility loci
【发表期刊】Nature Biotechnology
【发表日期】2024年5月9日
【作者及团队】Euler科技Yi Zhang联合武汉大学中南医院泌尿外科王行环团队
【IF】46.9
研究结果
一. 染色质可及性用于估计单细胞年龄
时钟样差异甲基化位点(ClockDML)的甲基化状态可能受染色质可及性影响,反之亦然,这种现象为推断细胞年龄提供了可能性。
作者确定了大量的ClockDML,这些位点在基因组中显示年龄相关的甲基化变化,这些位点的开放比例与细胞的年龄相关。
研究团队开发了一个名为EpiTrace的算法,可以从ATAC-seq数据中推断细胞的年龄。该算法准确预测了样本的年龄,显示出与已知发育轨迹的一致性。
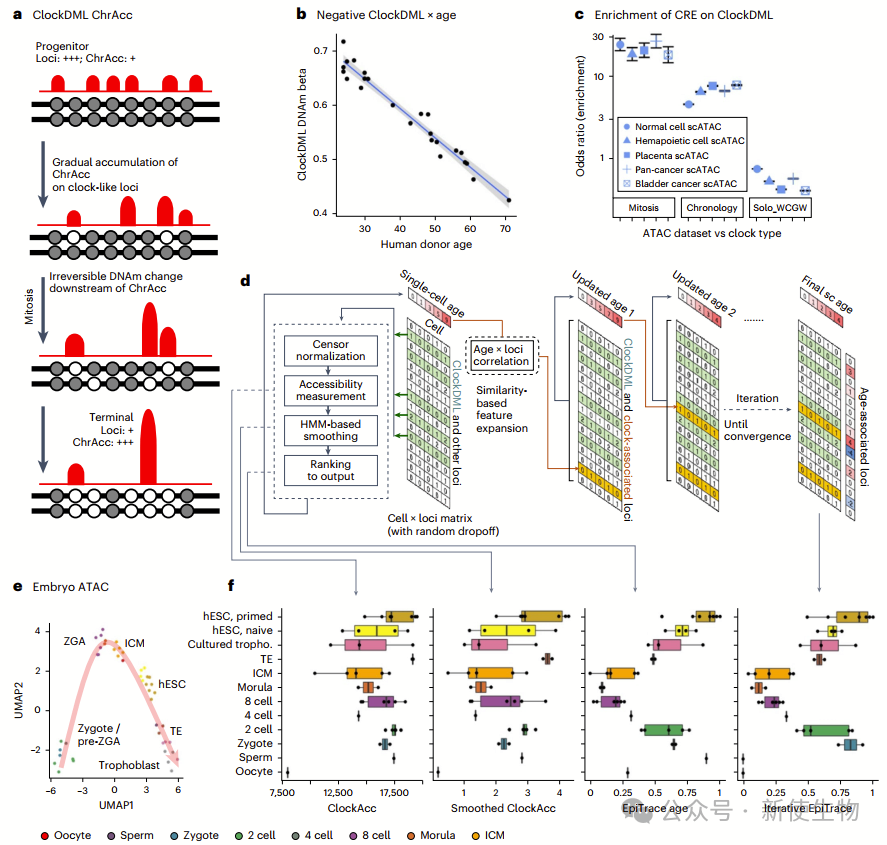
二. 跨细胞类型和动物物种推断细胞年龄
许多细胞和动物物种的ClockDML尚未确定。从人类PBMCs衍生的ClockDML可用于预测人类血细胞及非造血细胞的年龄。
作者将人类ClockDML映射到小鼠基因组,并使用这些参考位点计算小鼠scATAC-seq数据集的EpiTrace年龄。结果显示,使用人类ClockDML为参考的EpiTrace预测与使用小鼠ClockDML的预测结果非常接近。这表明EpiTrace可以跨不同物种使用ClockDML来预测单细胞年龄。
他们还将人类ClockDML映射到斑马鱼基因组,并使用它来预测斑马鱼的单细胞年龄,结果表明预测结果与已知的样本年龄密切相关。
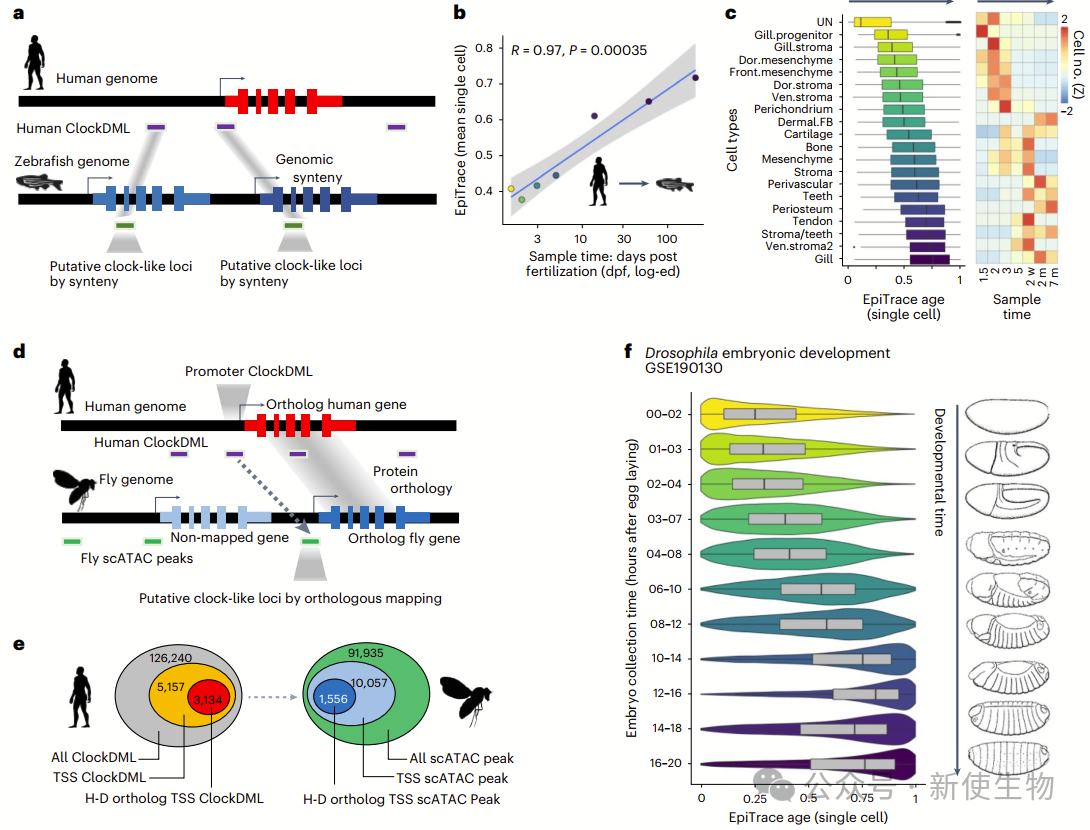
三. 染色质可及性的变化先于ClockDML上的DNA甲基化转变
果蝇缺乏典型DNA甲基转移酶,基因组中仅有很少的胞嘧啶甲基化。尽管如此,EpiTrace在果蝇基因组上的工作表明,钟摆式染色质可及性可能与DNA甲基化无关。
作者跟踪了染色质可及性和DNA甲基化的变化,发现染色质可及性变化先于DNA甲基化变化。通过实验证实,染色质可及性的改变足以导致DNA甲基化的变化。
此外,他们还验证了DNA甲基化的变化对染色质可及性的影响。这些结果表明,钟摆式染色质可及性在DNA甲基化变化之前发生,且与DNA甲基化无关。
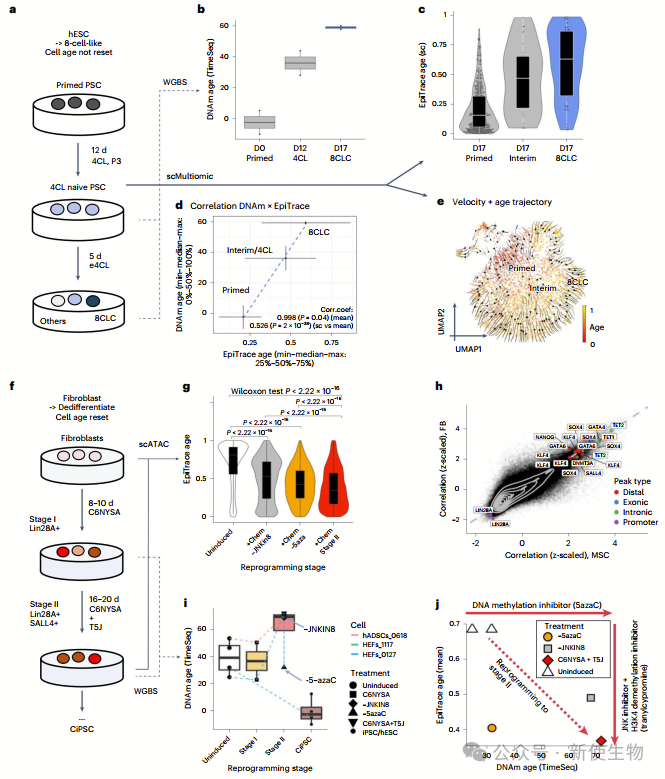
四. iPSC诱导过程中的表观年龄逆转
作者在人类胚胎干细胞培养物中进行了EpiTrace测试,这些细胞经历了重编程,从“4细胞样原始PSC”状态向“八细胞样”状态转变。
他们测量了单个细胞的EpiTrace年龄,并将其与同一培养物的全基因组亚硫酸盐测序(WGBS)预测的生物年龄进行了比较。结果显示,有丝分裂年龄随着细胞转变而增加,单个细胞的年龄逐渐增加。DNAm和EpiTrace的生物年龄估计高度相关,表明染色质可及性(ChrAcc)在ClockDML上能够准确预测单个细胞的生物年龄。
作者还发现EpiTrace年龄预测在诱导阶段II明显下降,表明这些细胞经历了“年轻化”。这些结果表明,ChrAcc上的EpiTrace能够独立于ClockDML的DNAm变化,准确预测细胞的生物年龄。
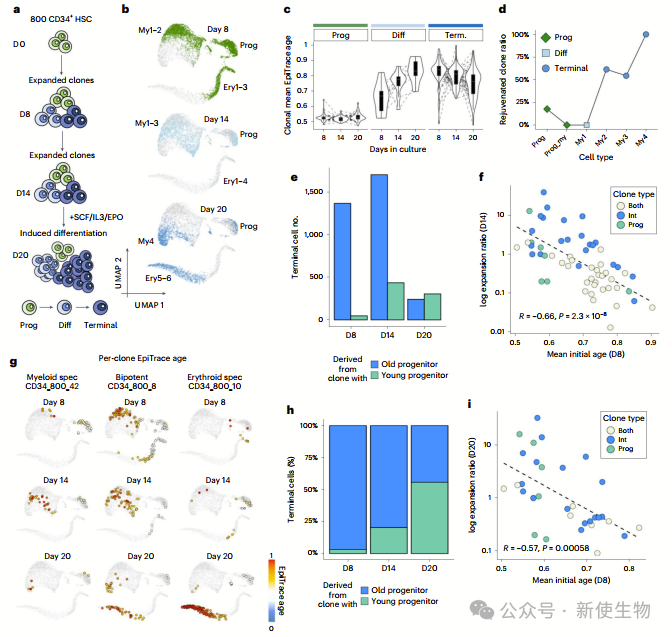
五. 表观遗传年龄决定细胞未来扩增潜力
团队利用一个线粒体增强的单细胞ATAC测序数据集对CD34造血干细胞进行了研究,它们在体外扩增14天后被分化成髓系细胞。
作者使用EpiTrace估计这些细胞的有丝分裂年龄,并发现这些年龄预测与已知的采样日高度一致,能够追踪同一克隆中细胞的年龄。
他们观察到,髓系细胞的年龄随着从前体到终端细胞的转变而增加,而强制分化会增加分化细胞的年龄,但会降低终端细胞的年龄。
作者的研究结果支持了这样一个模型:在体外HSC刺激扩张期间,终端分化的细胞更可能来自年轻的前体细胞,这些细胞具有更强的扩张和分化能力。
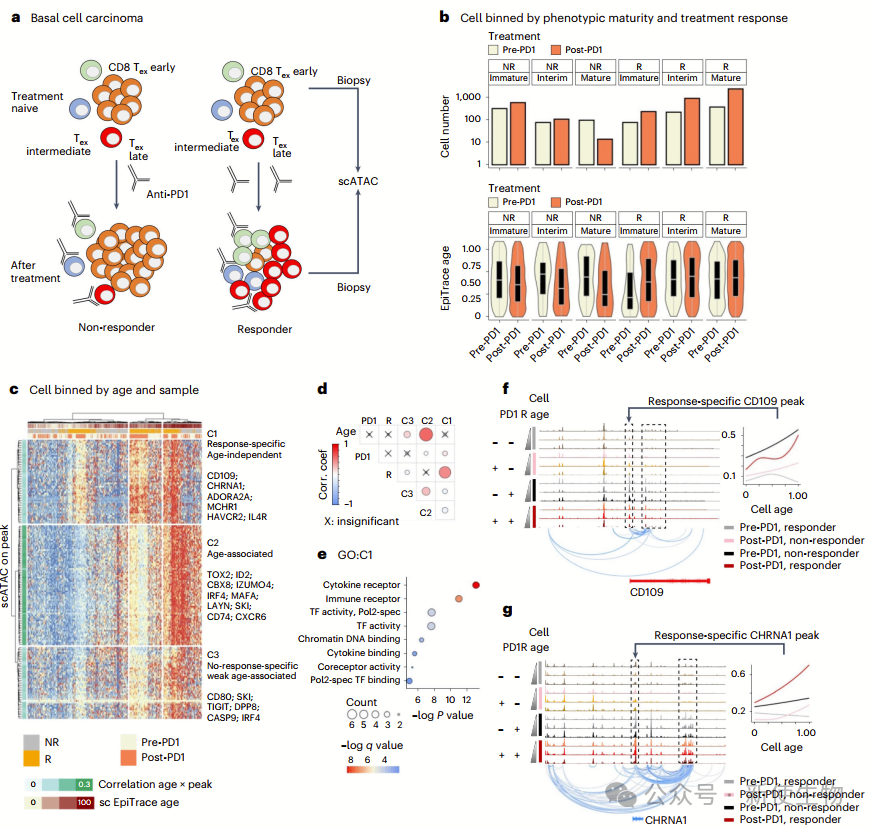
六. 阐明抗PD1反应的T细胞标志物
作者使用CD34数据集进行了体外实验,但为了在更复杂的体内环境中测试EpiTrace,作者转向了基底细胞癌组织的单细胞ATAC-seq数据集。结果表明,抗PD1治疗后,反应者的免疫细胞数量显著增加,而非反应者中没有。
通过分析染色体开放性与细胞年龄的相关性,他们确定了一些与抗PD1治疗响应相关的染色体区域,这些区域与免疫反应通路相关,这些发现为未来研究提供了有价值的线索。
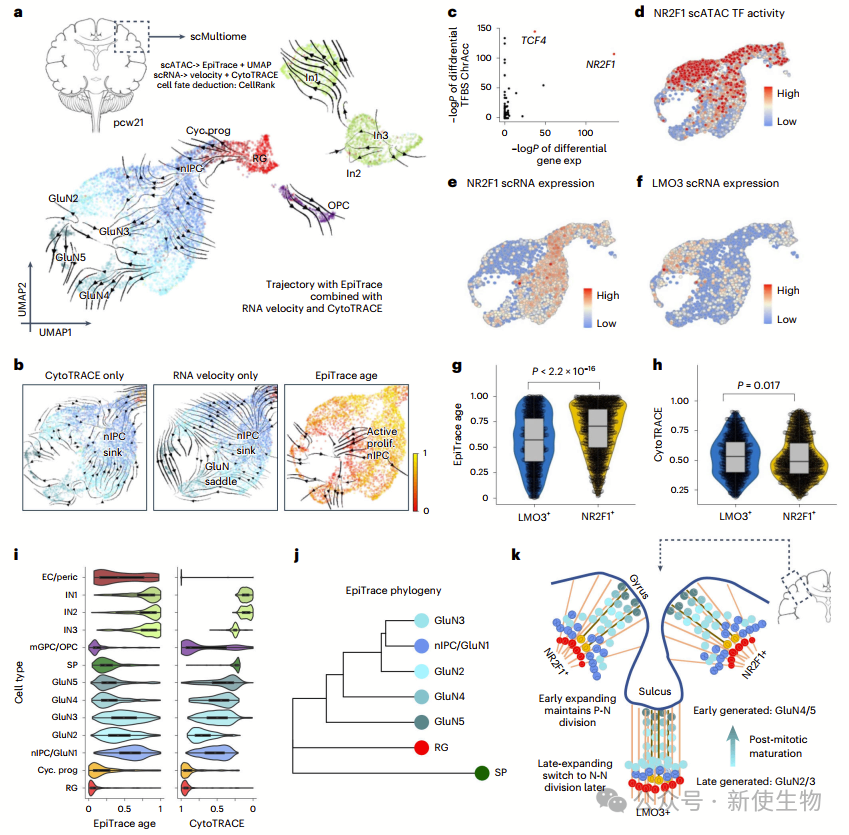
七. 揭示大脑皮层回折过程中的发育历史
团队利用EpiTrace分析了人类胚胎大脑皮层pcw 21的scMultiomic数据集,探究了谷氨酸能神经元(GluN)的发育轨迹。
GluNs从星形胶质(RG)发展成神经中间前体细胞(nIPCs),然后经历一系列成熟过程。作者发现EpiTrace年龄核揭示了正确的发展方向,而NR2F1转录因子在皮质回纹形成中起重要作用。
总结
scATAC-seq可重建发育轨迹,但推断轨迹具挑战。本文开发了EpiTrace,利用ClockDML的染色质可及性来确定细胞年龄,并进行谱系追踪。EpiTrace与发育级别一致,与DNAm时钟相关,且与突变谱系追踪、RNA速度和干细胞预测相辅相成。在scATAC-seq数据中应用EpiTrace揭示了多个领域的生物学见解。
新使生物(neoribo)推出国内首款超高分辨率翻译组建库试剂盒。
我们的核糖体印迹分析技术(QEZ-seq®)适用于哺乳动物、植物、真菌等多类物种,无需复杂操作,建库只需2小时。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
简便快速的操作让翻译组分析不再设有门槛,每位科研学者都能轻松上手。


了解更多翻译组咨询新使生物


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们