导读
研究学者们在各种RNA类型中已经发现了超过170种转录后修饰,它们在基因表达调控中发挥着多样的作用。其中,5-甲基胞嘧啶(m5C)是一种常见的RNA修饰,存在于rRNA、tRNA和mRNA中。
在mRNA中,m5C可以被特定识别蛋白(如ALYREF、YBX1、YBX2、FMRP和SRSF2)识别,并调控RNA代谢的各个方面,包括输出、稳定性、剪接和翻译。
全面了解RNA修饰需要准确和灵敏的检测技术。与m6A甲基化相比,m5C在mRNA中的修饰水平较低,需要强大的检测方法来区分真实信号和假阳性。目前最常用的m5C检测方法是RNA亚硫酸氢盐测序(BS-seq)。
在BS-seq中,未修饰的胞嘧啶(C)被转变为尿嘧啶(U),而m5C保持不变,从而标记未转换的C为m5C。然而,BS-seq面临以下挑战:
1)依赖C完全转变为U,结构化区域的转换不完全会导致假阳性
2)苛刻反应条件导致RNA降解,阻碍低输入样本和低丰度RNA的检测
3)C转变为U降低了序列复杂性,影响文库构建和测序质量。
这些限制使得对m5C的全面理解变得困难,特别是在mRNA中,不同研究对m5C位点的数量仍有争议。
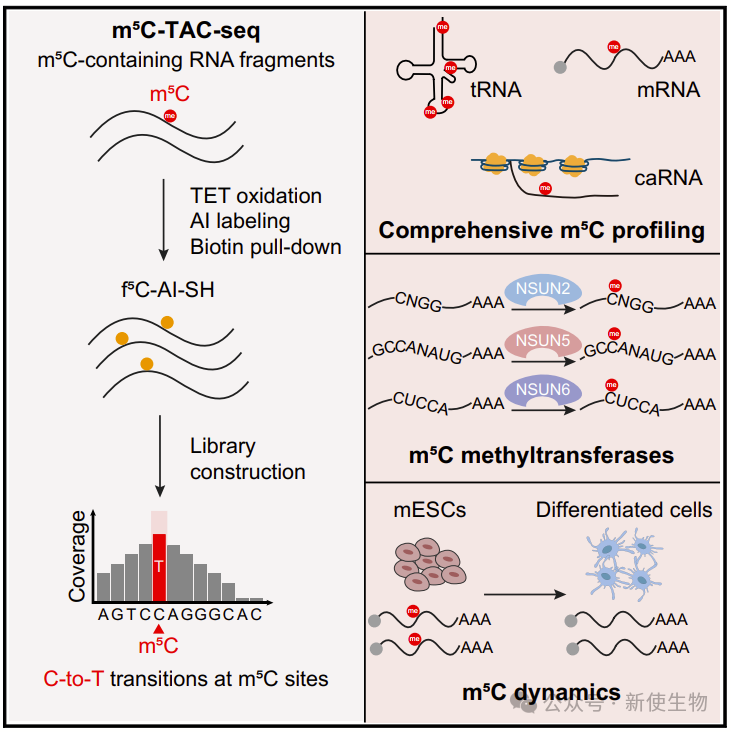
2024年7月12日,浙江大学医学院李笑雨与北京大学生命科学学院伊成器团队在Molecular Cell上发表了一篇题为“Base-resolution m5C profiling across the mammalian transcriptome by bisulfite-free enzyme-assisted chemical labeling approach”的论文,文章开发了一种无BS、碱基分辨率的m5C检测方法——m5C-TAC-seq(TET辅助化学标记)。该方法对RNA温和,允许在低丰度和低序列复杂性的RNA中直接检测m5C,可用于研究不同RNA物种和生物学背景下的m5C甲基化。

文章索引
【标题】Base-resolution m5C profiling across the mammalian transcriptome by bisulfite-free enzyme-assisted chemical labeling approach
【发表期刊】Nature Biotechnology
【发表日期】2024年7月12日
【作者及团队】浙江大学医学院李笑雨与北京大学生命科学学院伊成器团队
【IF】46.9
研究结果
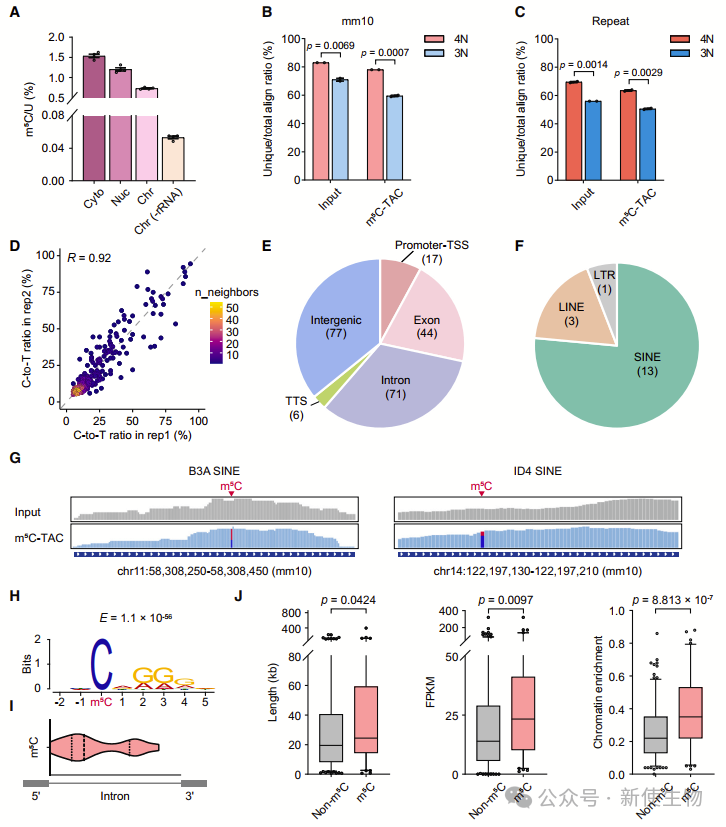
一. TET辅助选择性化学标记实现了稳健且灵敏的RNA m5C检测
在之前的研究中,作者展示了AI可选择性标记DNA中的5fC,实现高效的C到T转换,用于全基因组5fC分析。研究团队设想TET能将RNA中的m5C氧化为f5C,并用AI标记以引发C到T转换,实现转录组范围内的m5C检测。
实验结果显示,AI标记高效温和,能实现f5C RNA的30倍富集,并诱导高C到T转换率(93%-98%)。验证表明,TET酶可以将RNA m5C氧化为f5C,AI标记几乎标记了所有新生成的f5C(99%),且保持RNA完整性。
总体而言,m5C-TAC策略灵敏、特异,对RNA影响较小,适用于稳定的m5C检测。
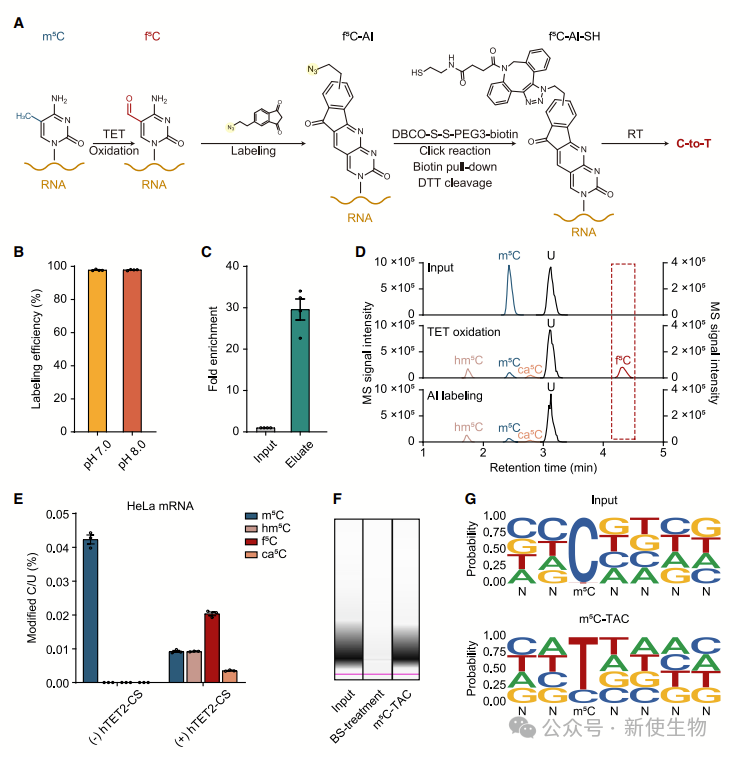
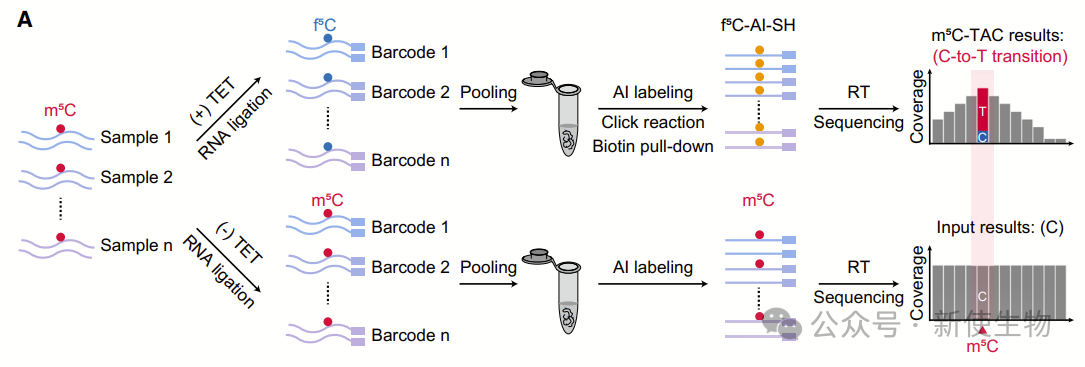
二. m5C-TAC-seq实现了半定量和基因组分辨率的RNA m5C分析
作者在转录组水平评估了其应用,并采用多重文库构建策略,减少起始材料使用量和样本间技术差异。
通过混合不同比例的未修饰C和完全修饰m5C进行m5C-TAC-seq,结果显示即使在2.5%甲基化水平下,m5C-TAC-seq仍能可靠地区分m5C和未修饰C。这一方法能够半定量地揭示m5C在多种生物过程中的动态变化。
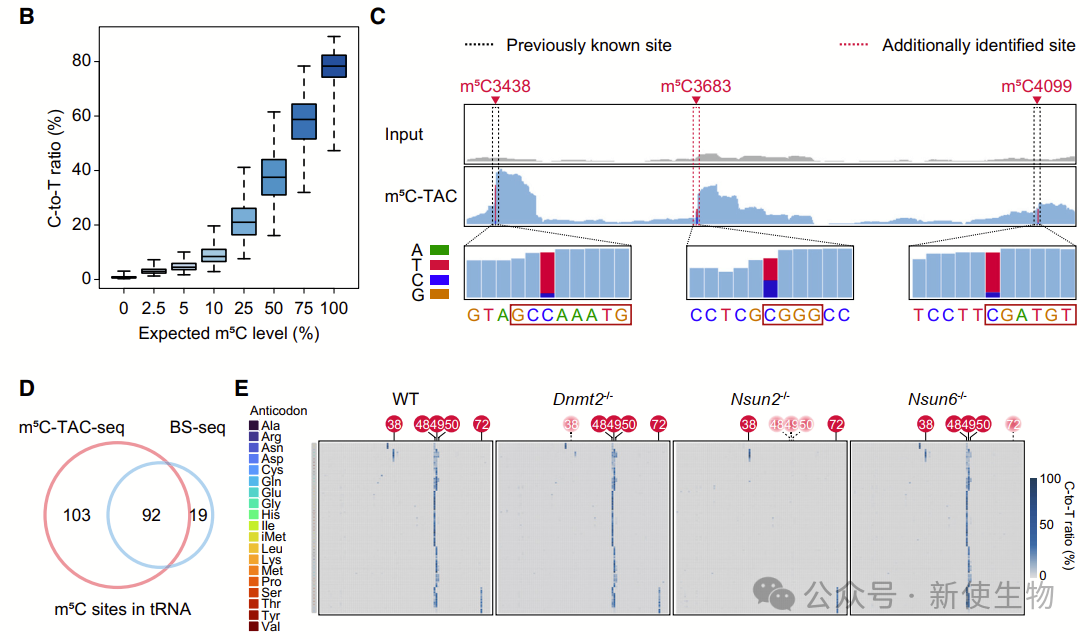
三. m5C-TAC-seq揭示了rRNA和小非编码RNA中的额外m5C位点
作者利用m5C-TAC-seq分析了mESCs中的tRNA和rRNA,发现了200个共享的m5C位点,证明其高可重复性。
在rRNA中,他们确认了两个已知的m5C位点,并额外发现了一个新的m5C3683位点。通过基因敲除验证,确认了NSUN2在m5C3683的甲基化中的作用。
此外,m5C-TAC-seq还成功识别了在低丰度非编码RNA中的m5C位点,并确定了它们的甲基转移酶。
四. 人类转录组中基本分辨率的全面m5C景观
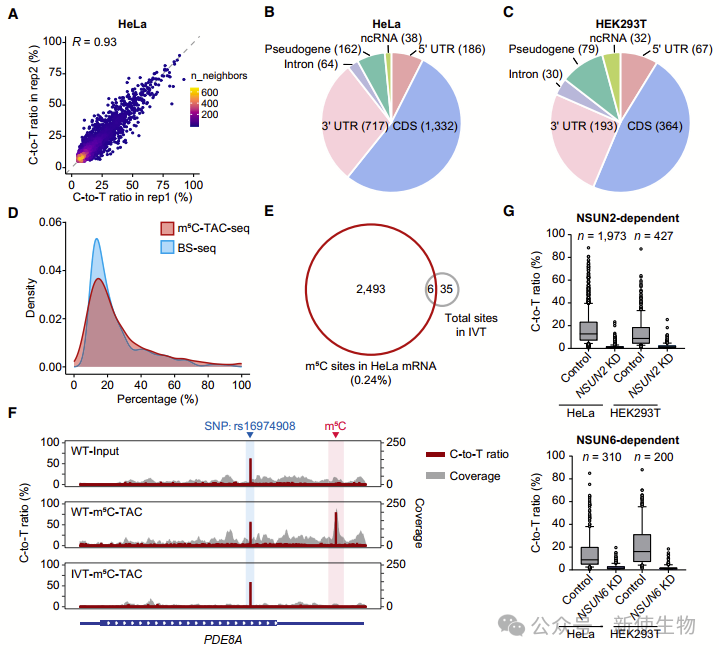
在HeLa和HEK293T细胞中应用m5C-TAC-seq,作者检测到大约2,499个和765个高度可复制的m5C位点。这些位点主要分布在mRNA的编码序列(CDS)和3'端非翻译区(3' UTR),显示出该方法的高精度和可靠性。
与BS-seq相比,m5C-TAC-seq不仅展示了更高的重现性和唯一映射比率,还能够检测到超过1,500个额外的m5C位点,尤其是那些低量程度的位点。
进一步的分析表明,m5C-TAC-seq对于mRNA m5C检测的稳健性和准确性,尤其是在酶依赖位点的鉴定上具有显著优势。
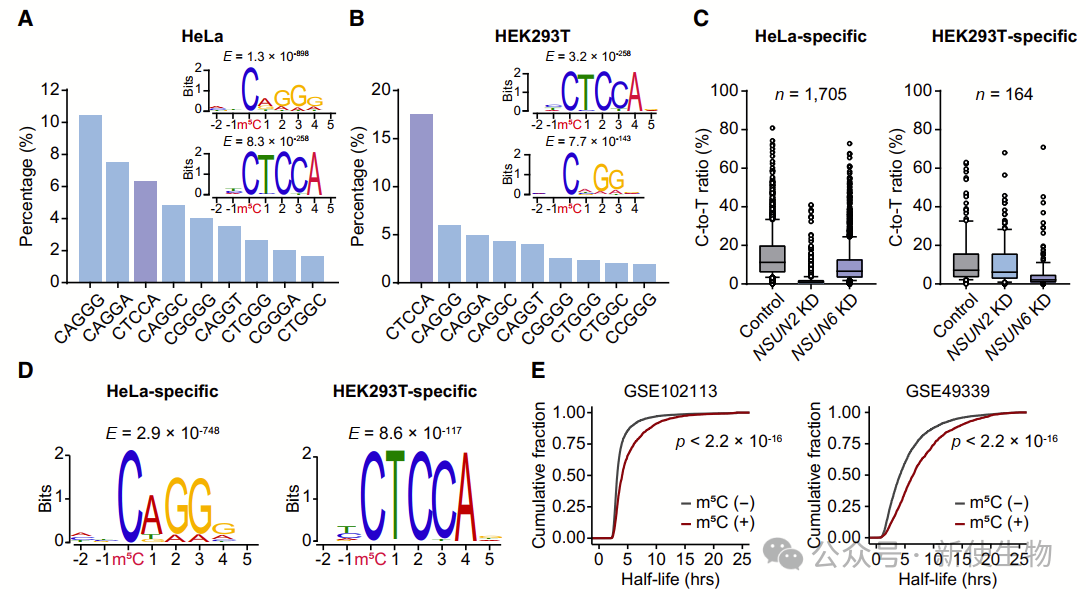
五. HeLa和HEK293T细胞之间的差异m5C甲基组
作者比较了HeLa和HEK293T细胞的m5C甲基组。
总体上,HeLa细胞中鉴定的m5C位点显著多于HEK293T,且这些位点主要由NSUN2和NSUN6催化。HeLa细胞特有的m5C位点表现出NSUN2依赖性,而HEK293T细胞特有的位点则表现出NSUN6依赖性。
进一步分析表明,m5C甲基化在RNA稳定性和翻译调控中起重要作用。
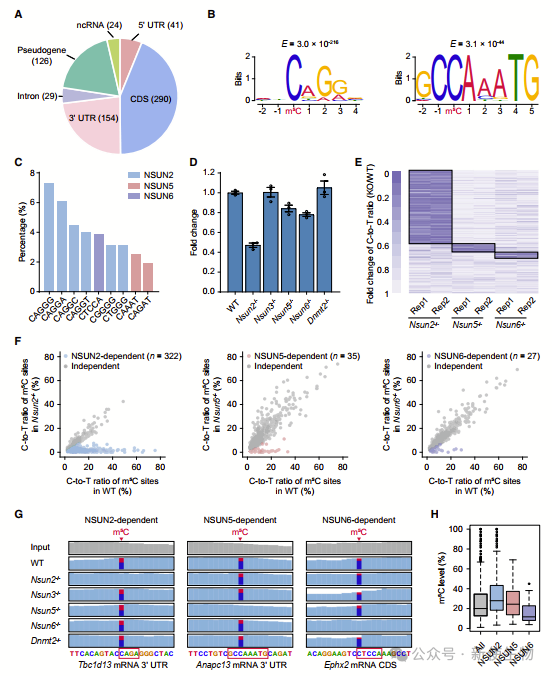
六. m5C甲基转移酶依赖的mESC m5C甲基组
在mESC mRNA中进行了m5C-TAC-seq,发现了664个m5C位点,验证了其准确性和灵敏度。
主要的m5C甲基转移酶包括NSUN2、NSUN5和NSUN6,分别催化了322、35和27个m5C位点。这些酶依赖位点在CDS和3'UTR区域中均有富集,突显了NSUN5的作用,并显示了m5C-TAC-seq在分析m5C动态变化中的可靠性。
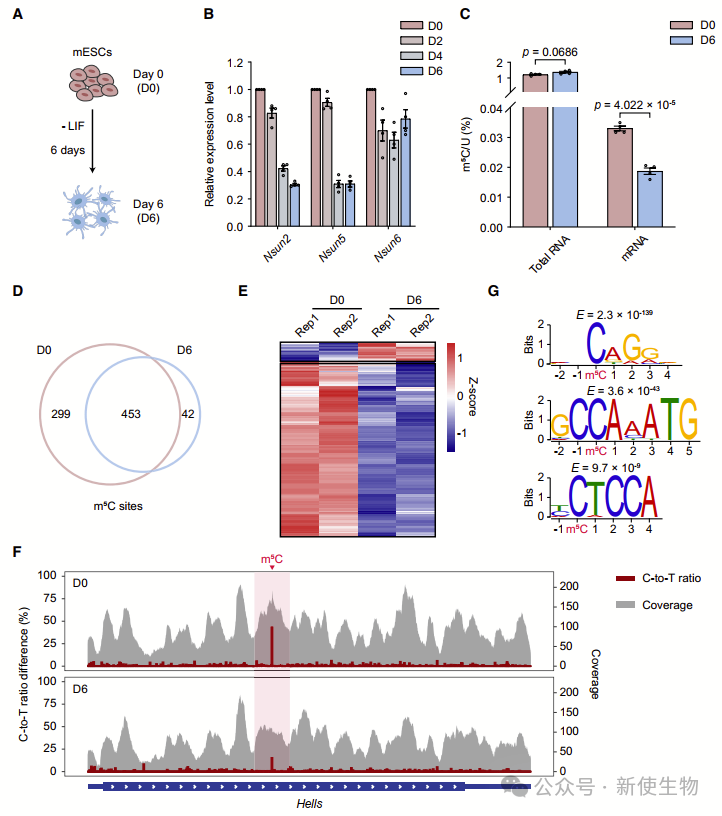
七. mESC分化过程中动态调控的m5C甲基组
mESC分化中m5C甲基组动态调控研究发现,随着分化过程,Nsun2和Nsun5表达显著下降,Nsun6略有减少。
质谱分析显示mRNA的m5C甲基化水平减少约50%。m5C-TAC-seq进一步证实,大多数m5C位点在分化中呈现下调状态,涵盖421个下调位点和45个上调位点。
这些变化与相关基因表达水平的下降相对应,尤其是与细胞周期和分裂相关的基因。这些结果提示,动态调节的mRNA m5C甲基组可能对mESC的细胞命运决定起重要作用。
八. 共转录的caRNA中m5C沉积
作者通过质谱发现caRNA中存在m5C。由于重复序列和低丰度问题,采用m5C-TAC-seq鉴定出215个m5C位点,主要位于基因间区和内含子区域。m5C位点倾向于m5CNGG模体,暗示NSUN2可能在caRNA中起作用。这表明m5C可以在caRNA中沉积,增加了调控层次。
总结
本文开发的m5C-TAC-seq,无需重亚硫酸盐,通过TET辅助氧化和化学标记实现碱基分辨率的m5C检测。该方法识别了大量人类和小鼠细胞中的m5C位点,确定了大多数m5C位点的甲基转移酶,包括NSUN5在mRNA m5C沉积中的作用,并揭示了mESC分化过程中m5C的动态变化。
新使生物(neoribo)推出国内首款超高分辨率翻译组Ribo-seq建库试剂盒。
我们的Ribo-seq核糖体印迹分析技术(QEZ-seq®)适用于哺乳动物、植物、真菌等多类物种,无需复杂操作,建库只需2小时。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
简便快速的操作让翻译组分析不再设有门槛,每位科研学者都能轻松上手。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可联系杭州新使生物

关注新使生物,了解更多翻译组前沿信息


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们