导读
在真核生物中,基因表达涉及多个细胞区室。DNA在细胞核中转录后,mRNA前体经过加帽、剪接、3′ 端截短及poly(A) 添加,形成成熟mRNA,经核输出后在细胞质中翻译。mRNA稳定性受5′或3′ UTR调控,如5′ UTR上的uORF可通过核糖体停滞诱导mRNA降解,同时降低主ORF翻译效率。
转录与mRNA降解原被视为独立过程,现研究表明二者存在反馈调控:
A)斑马鱼和小鼠研究发现,由提前终止密码子(PTC)引发的无义介导的mRNA降解(NMD)可上调功能相关基因转录,mRNA片段可能通过染色质修饰触发遗传补偿反应。
B)酵母研究显示,mRNA降解因子突变可增加mRNA稳定性,但不影响其稳态水平,表明mRNA半衰期变化可通过转录反向调节补偿。
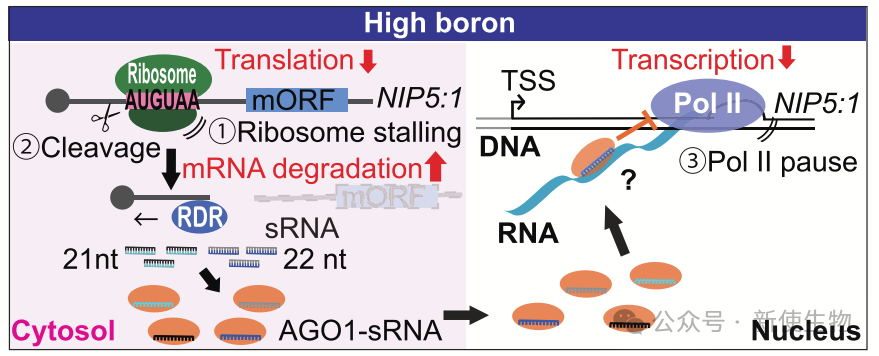
硼(B)是植物生长所必需的营养元素,但过量时具有毒性。NIP5;1作为硼酸转运蛋白,在低硼条件下促进吸收,而在高硼条件下mRNA不稳定,以防止过量吸收。该降解受5′ UTR上AUGUAA诱导的核糖体停滞触发,硼稳定eRF1并促进其与Met-tRNAi互作,阻碍翻译再起始,引发mRNA切割与降解。此外,NIP5;1表达受STOP1调控,但未发现其对硼的响应,硼依赖的NIP5;1转录调控机制仍待研究。
2025年3月24日,东京大学农业与生命科学研究院Toru Fujiwara团队在Nucleic Acids Research上发表了一篇题为“Ribosome stalling-induced NIP5;1 mRNA decay triggers ARGONAUTE1-dependent transcription downregulation”的论文,提出了一种新的调控模型,即翻译过程不仅感知养分状态以选择性降解mRNA并减少翻译,而且该降解过程还能与AGO1共同作为触发信号,诱导转录下调。这些过程相互交织,形成转录、mRNA降解与翻译之间的多层次耦合,实现高度精细的调控并对环境变化作出迅速响应。

索引
【IF】16.97
研究结果
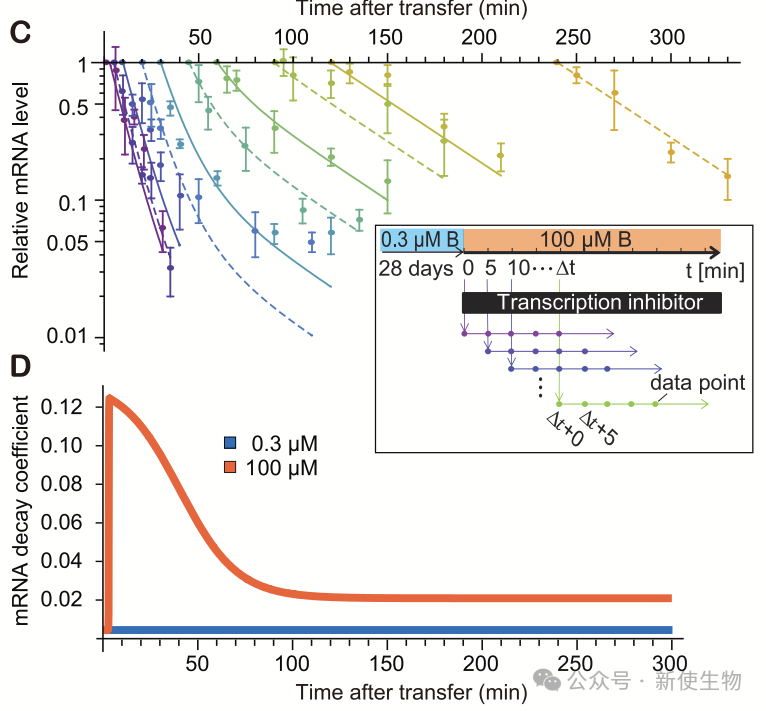
一. NIP5;1 mRNA的下调具有硼浓度依赖性
研究发现NIP5;1 mRNA在低硼条件下积累显著高于高硼条件。不同硼浓度下,NIP5;1 mRNA随浓度增加而逐渐下调,并在90分钟内达到新的稳态,表明其下调具有浓度依赖性。

二. 仅靠mRNA降解无法解释观察到的NIP5;1 mRNA动态
硼浓度变化不仅通过uORF触发mRNA降解,还会通过调节转录速率影响NIP5;1 mRNA的下调。
在低硼浓度下,mRNA降解速率较低,转录速率较高,而在高硼浓度下,mRNA降解速率增加,转录速率降低,导致NIP5;1 mRNA的积累减少。
三. NIP5;1 mRNA转录在硼响应下被下调
30 μM硼下mRNA前体减少至62%,100 μM硼下转录活性下降46%。转移至100 μM硼后,mRNA前体在30分钟内开始下降,120分钟达到稳态,表明硼通过转录下调NIP5;1表达。
四. ATGTAA序列在硼依赖性转录调控中是必需的
在实验中,含ATGTAA的转基因植物在硼浓度变化下表现出转录下调,而缺失ATGTAA的突变体未出现这种变化。
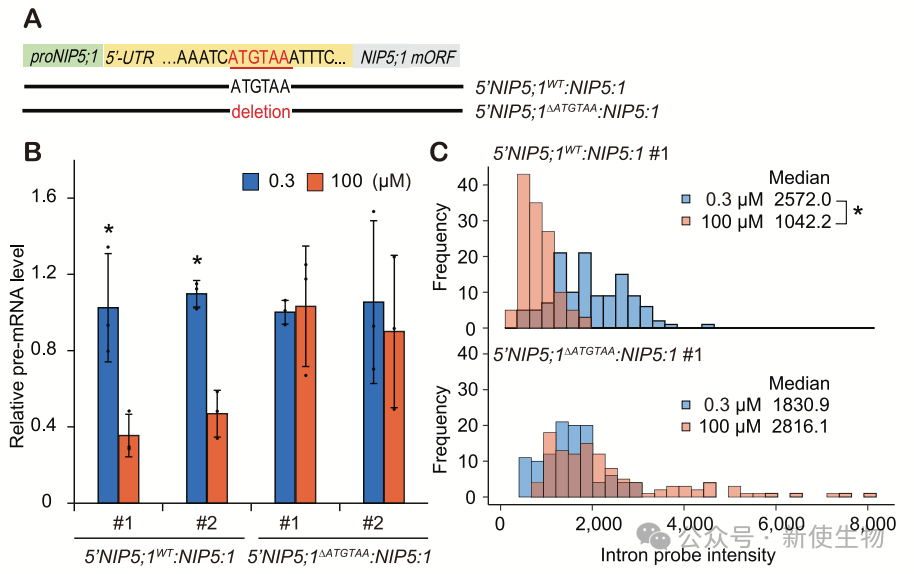
五. 转录下调在转基因中起作用
作者研究发现NIP5;1转录下调通过mRNA降解中间体在转式调控中起作用。具体来说,ATGTAA序列介导了含AUGUAA序列的mRNA降解,这一过程依赖于XRN4酶的作用。
实验表明,XRN4突变会稳定这些降解中间体,并导致NIP5;1转录下降调。进一步的分析也支持这一结果,表明mRNA降解中间体的积累是转录下调的关键因素。
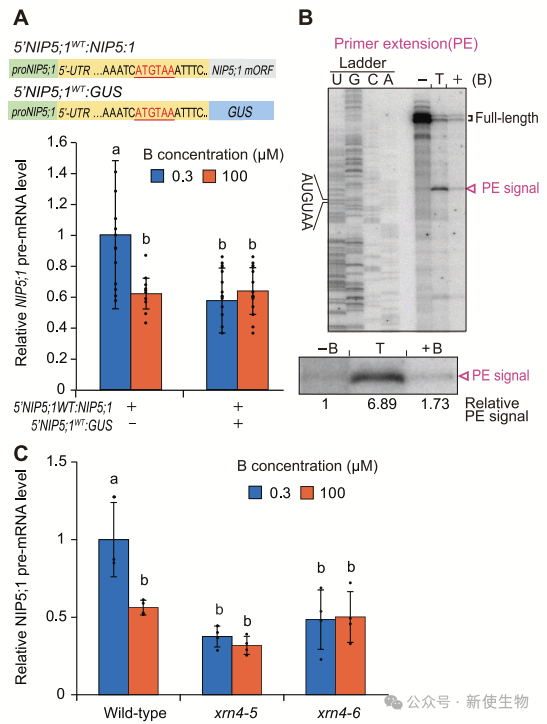
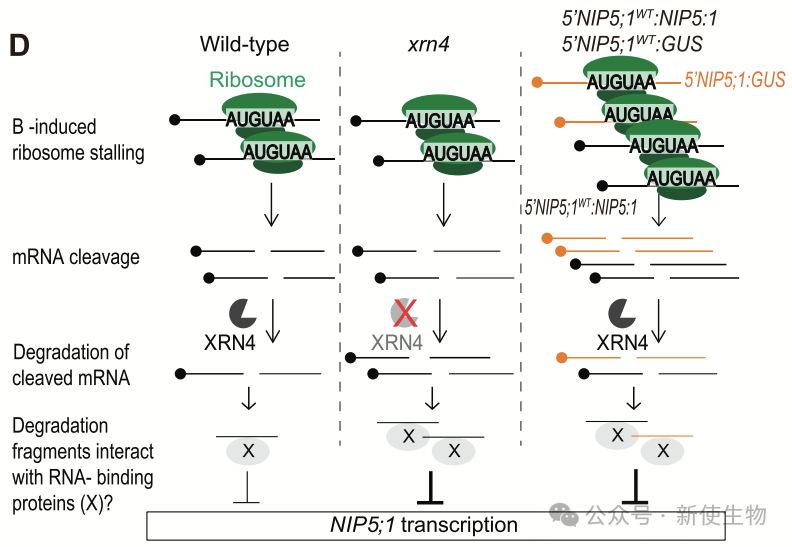
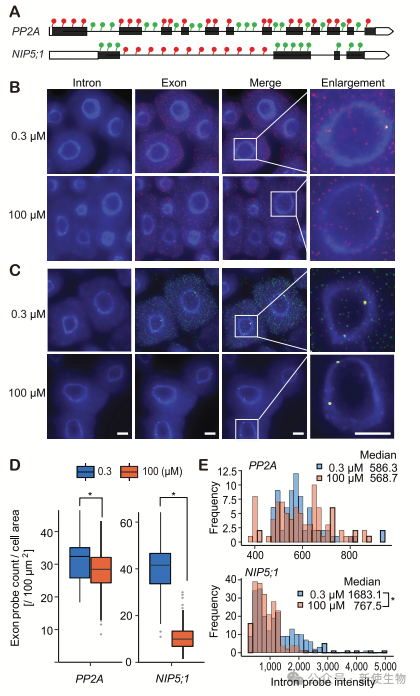
六. sRNA-seq鉴定出可能源自NIP5;1降解中间体的AGO相关小RNA
NIP5;1 mRNA降解中间体缺乏5′帽或poly(A)尾时,可以被RNA依赖的RNA聚合酶处理为双链RNA(dsRNA),进而产生小RNA(sRNA)。
通过分析高通量测序数据,作者发现NIP5;1 5′-UTR区域的21–22 nt sRNA在转录本中的正链或反链上富集,这些sRNA序列与NIP5;1 mRNA降解片段相关,支持它们源自NIP5;1 mRNA的降解中间体。
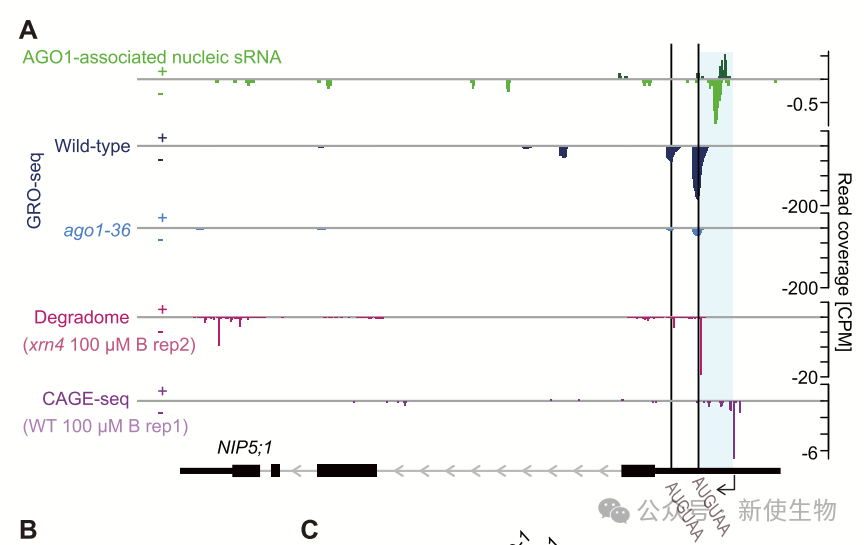
七. NIP5;1 mRNA的内切降解产生了截短的5′片段
AGO1相关的小RNA分析表明,小RNA来源于NIP5;1 mRNA的5′片段。通过3′ RACE实验发现,5′片段和3′片段是通过内切降解产生的,且二者在切割位点上下游相连。
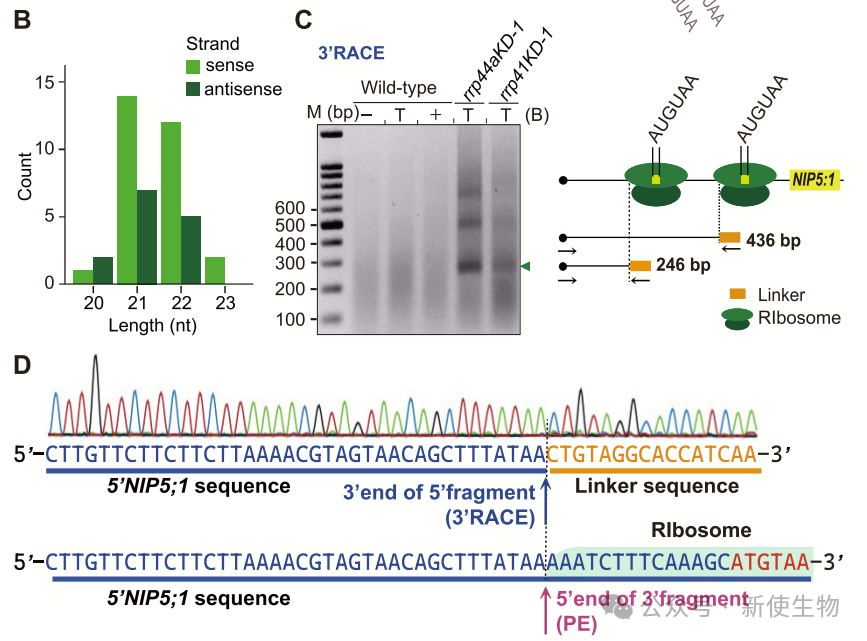
八. AGO1在NIP5;1 5′-UTR上调控RNA聚合酶暂停
在野生型植物中,NIP5;1的5′-UTR区域出现转录暂停峰值,且这些峰值在ago1突变体中减弱,表明AGO1参与转录暂停。
此外,ago1突变体中B依赖的转录下调完全消失,进一步证明了AGO1对NIP5;1转录下调的必需性。
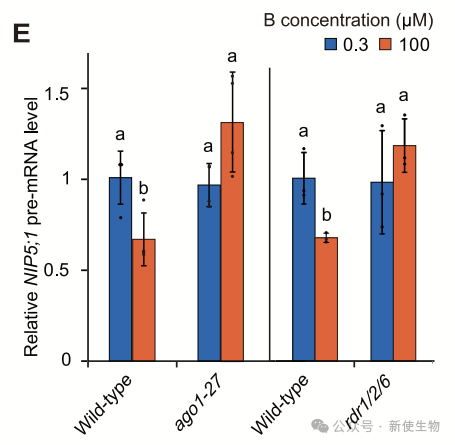
九. RDRs在B依赖的NIP5;1转录下调中是必需的
sRNA与NIP5;1 mRNA的正反义链序列相同,表明dsRNA通过RDR途径从mRNA降解中间体合成。
研究表明,B依赖的NIP5;1转录下调依赖于RDRs,且AGO1和RDRs共同作用于sRNA介导的转录下调机制。sRNA通过RDR途径从NIP5;1降解中间体产生,结合AGO复合物,诱导RNA Pol II在5′ UTR区域停顿。
总结
本文揭示了拟南芥NIP5;1基因表达的多层次调控机制。B依赖的NIP5;1 mRNA降解由5′-非翻译区的AUGUAA序列上的核糖体停顿触发。小RNA和AGO1在B依赖的转录下调中发挥重要作用,核糖体停顿、mRNA降解和转录下调共同调控NIP5;1的表达。这些发现表明类似的机制可能在其他真核生物中也存在。
我们能够针对微量细胞或组织,如卵母细胞、卵巢、临床穿刺样品等产出高质量翻译组数据结果。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可登录 www.neoribo.com
点击图片查看
点击图片查看


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们