导读
进化赋予了大量酶重新排列和多样化基因组的功能。这使得新基因的出现、免疫系统的发展以及病毒和移动遗传元件(MGEs)的传播成为可能。MGEs广泛存在,通常通过转座酶、整合酶、自剪切内切酶或重组酶进行移动,识别DNA的方式从位点特异性(如Cre和Bxb1重组酶)到半随机(如Tn5和PiggyBac转座酶)不等。
插入序列(IS)元件是简单的MGEs,广泛存在于细菌和古细菌中,通常使用自编码转座酶识别末端倒置重复序列(TIRs)。IS元件分为约28个家族,主要根据转座酶的催化残基分组,如DDE、DEDD等。
IS110家族元件是剪切和粘贴类型的MGEs,通过无痕切除和环化转座,使用DEDD催化基序,与RuvC解旋酶同源,缺乏TIRs,通常靶向微生物基因组中的重复元件。尽管IS110元件的DNA识别和重组机制尚不清楚,但先前的研究表明,重组酶开放阅读框两侧的非编码末端调节重组酶的表达。
2024年6月26日,加州大学伯克利分校生物工程系Patrick D. Hsu团队在Nature上发表了一篇题为“Bridge RNAs direct programmable recombination of target and donor DNA”的论文,文章展示了IS110环状形式驱动一种非编码RNA(ncRNA)的表达,该RNA具有两个结合环,分别识别IS110的DNA供体和插入靶位点,通过桥接供体和靶DNA,促进IS110重组酶的DNA重组。桥式RNA的结合环可以重新编程,以结合和重组不同的DNA序列,从而实现序列特异性的插入、倒位和切除。

值得注意的是,Patrick D. Hsu团队在2024年6月26日同日又发表了一篇Nature,通过解析桥RNA引导重组的结构,详细解释了其作用机制,进入公众号可查看此文章解读。

文章索引
【标题】Bridge RNAs direct programmable recombination of target and donor DNA
【发表期刊】Nature
【发表日期】2024年6月26日
【作者及团队】加州大学伯克利分校生物工程系Patrick D. Hsu团队
【IF】64.8
研究结果
一. IS621重组酶与一种非编码RNA(ncRNA)结合
IS110元件编码300-460个氨基酸的重组酶,具有DEDD RuvC样结构域和保守丝氨酸残基。这些重组酶通过无痕切除生成环状双链DNA,并插入特定基因组序列。
研究发现,IS110元件的切除重建了一个启动子,驱动ncRNA的表达。实验显示,IS621重组酶以高亲和力结合LE编码的ncRNA,这表明ncRNA在重组过程中发挥重要作用。
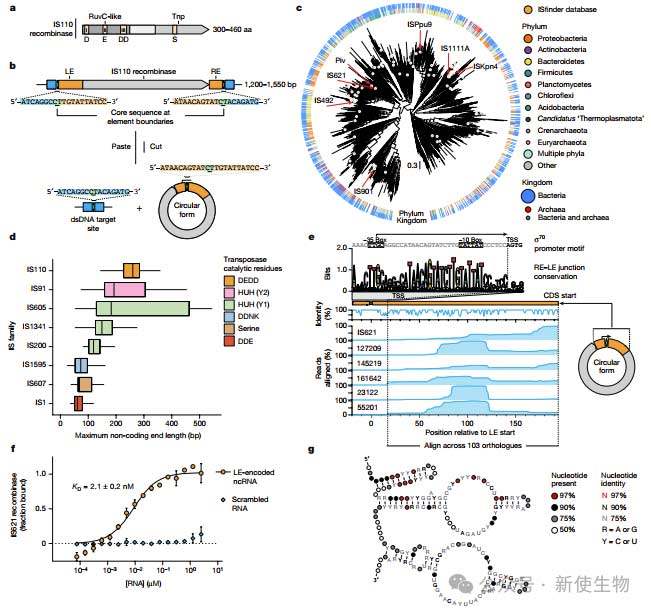
二. 非编码RNA(ncRNA)与靶DNA和供体DNA协调变化
作者分析了103个不同同源物的ncRNA共识次级结构,发现它包含一个5'茎环和两个内部环。第一个内部环变化较大,第二个内部环保守性较高。他们发现这些ncRNA可能与IS110元件的靶位点和供体DNA进行特定的碱基配对。
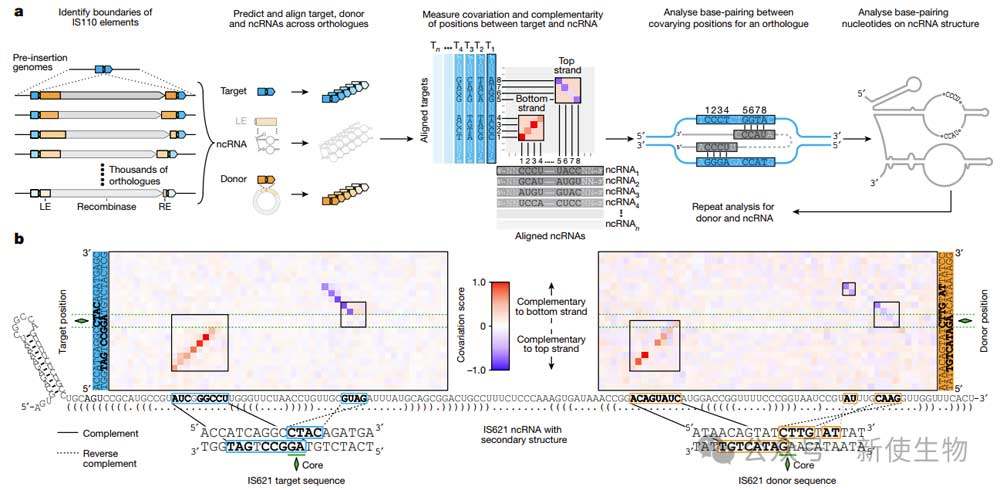
三. IS621 ncRNA连接靶位点和供体DNA
先前的研究仅在IS110宿主生物中成功,尚未在体外实现IS110元件的重组,作者发现ncRNA是体外重组的必需组分。
通过结合体外转录的ncRNA、纯化的IS621重组酶和靶位点及供体DNA序列的寡核苷酸,研究团队成功实现了重组。
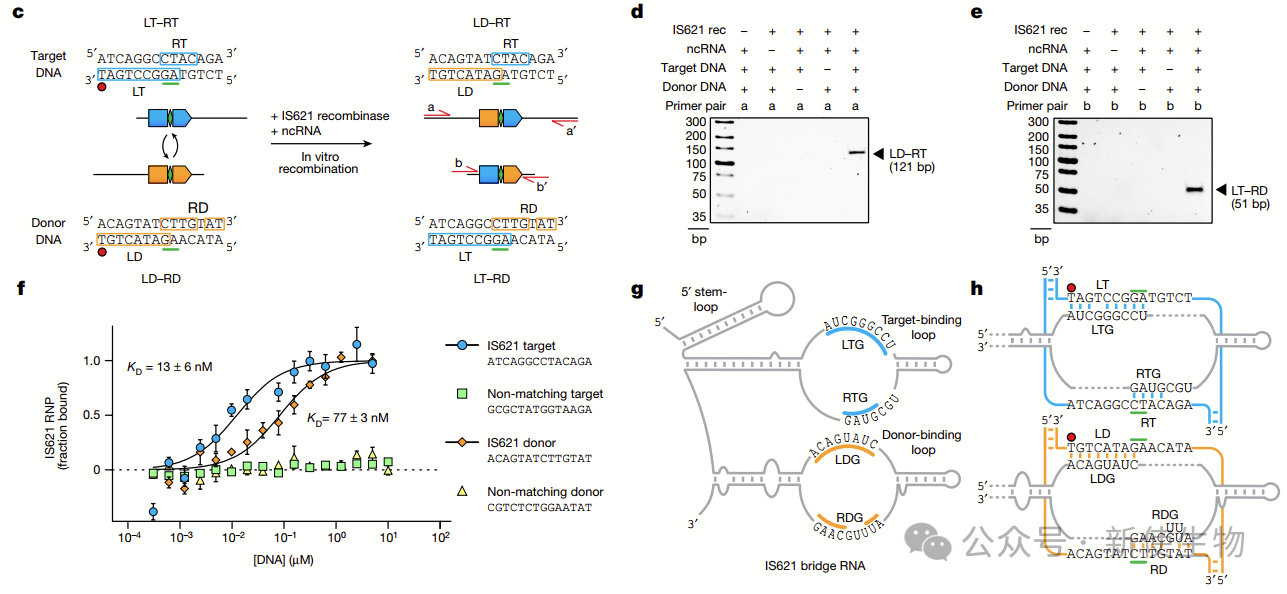
四. 可编程靶位点选择
先前的研究仅在IS110宿主生物中成功,尚未在体外实现其重组。作者发现桥接RNA是体外重组所需的关键组分。
通过在大肠杆菌中建立双质粒重组系统,他们验证了桥接RNA在重组中的作用。使用WT IS621供体和靶位点,团队成功观察到了预期的重组产物。
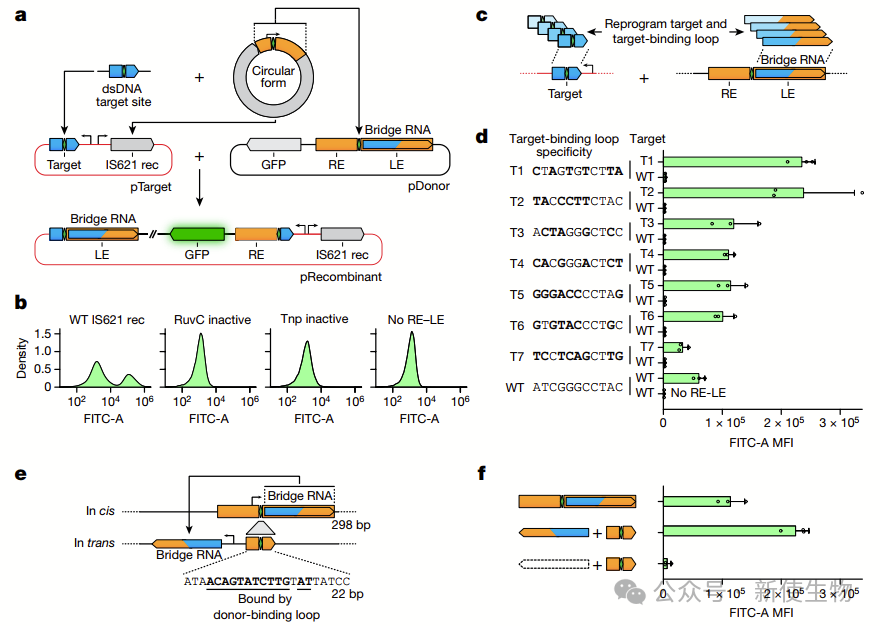
进一步的实验表明,通过重新设计桥接RNA的靶结合环,作者可以实现对靶位点选择的编程,从而在不同靶序列上实现高效的重组。
他们还展示了桥RNA可以通过转录方式表达,并增强其在重组中的效率。通过详细的错配耐受性和重新编程规则评估,确认了桥接RNA在靶位点特异性重组中的可编程性和精确性。
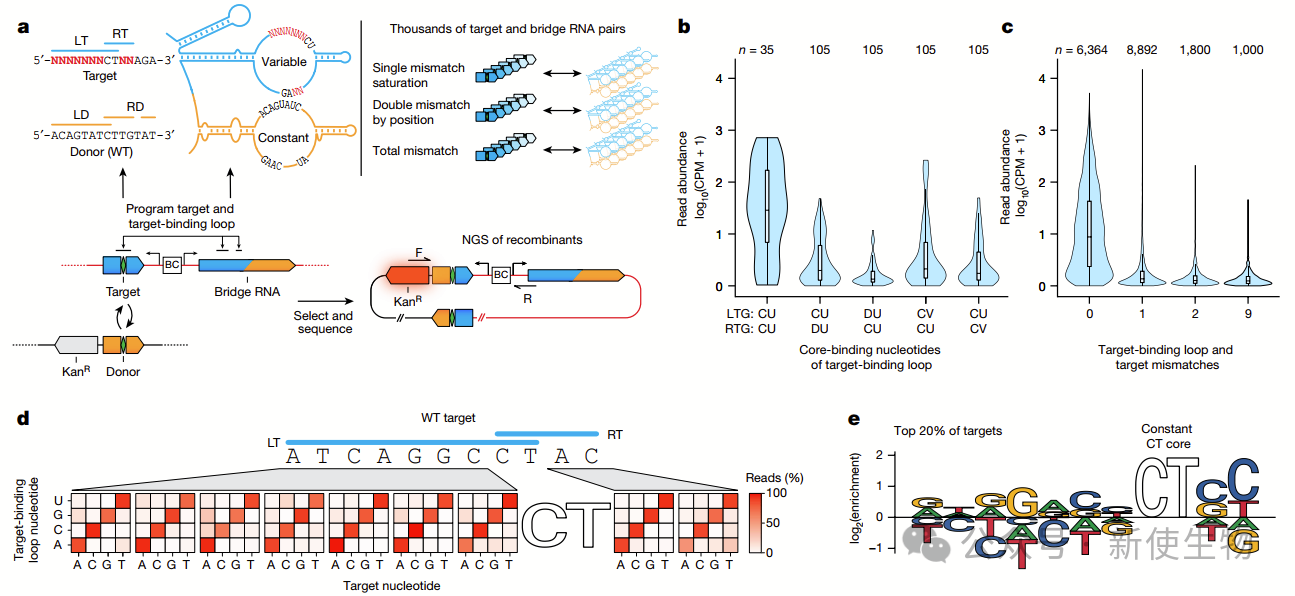
五. 可编程插入E. coli 基因组
使用WT IS621和桥RNA,作者在E. coli基因组中插入了一个带有22-bp WT供体序列的质粒。
他们检测到了173个独特的插入位点,其中144个位点位于已知的IS621靶向的REP元件内。几乎所有插入位点(96.21%)与自然环境中观察到的目标序列或桥接RNA编码的特异性高度吻合。
通过引入延伸的RTG序列,作者显著提高了插入到目标位点的特异性,并减少了非靶位点的插入。
此外,作者还观察到少量基因组内部的删除和倒转事件。这些实验结果展示了IS621在选择性插入特定位点和揭示其重组机制方面的强大能力。
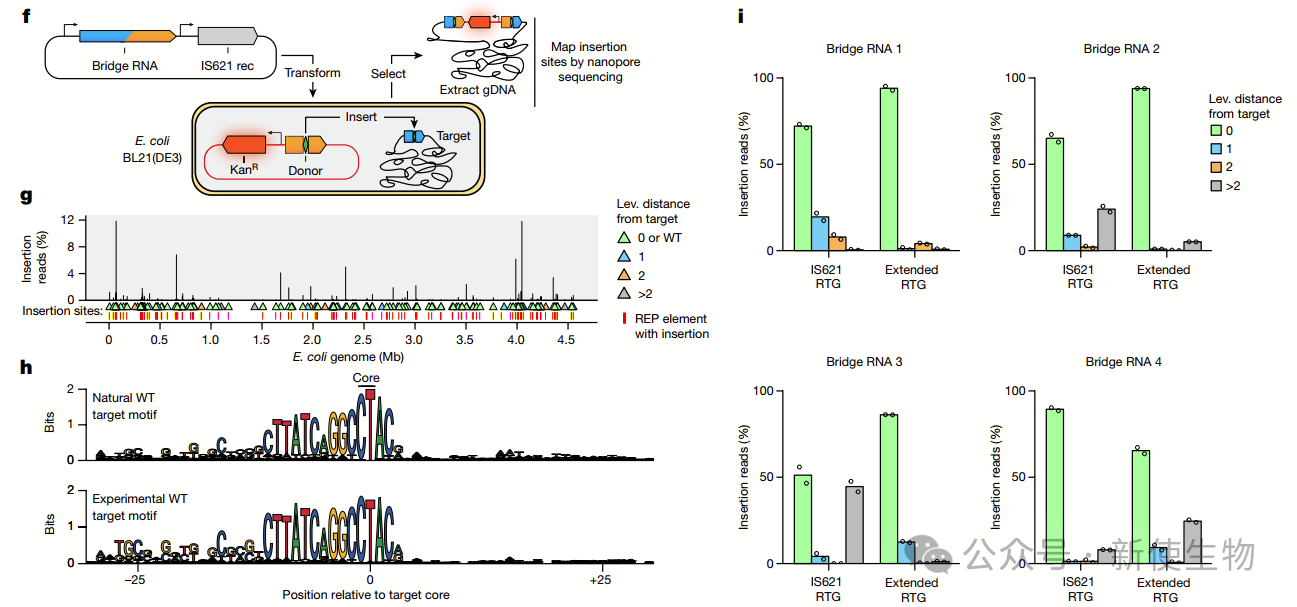
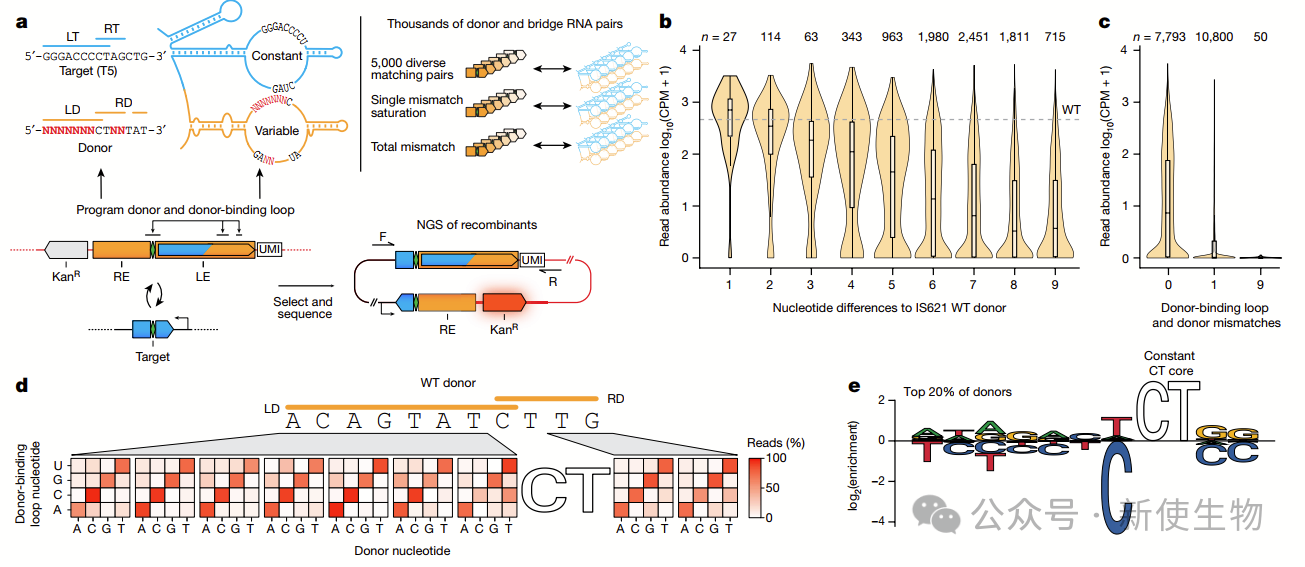
六. 编程桥接RNA的供体特异性
IS621元素中,供体序列比基因组目标序列更保守,表明供体结合环不容易像目标结合环那样重编程。
作者设计了一个供体特异性筛选系统,发现供体序列可以完全重编程。这表明桥接RNA允许模块化地重编程目标和供体DNA识别。
七. 可编程的DNA重排
除了用于DNA插入,像Cre这样的重组酶通常还用于DNA序列的切除或倒转。预先安装loxP识别位点可实现位点同向切除和异向倒转。
IS621重组酶据推测能够实现可编程的切除和倒转,展示了控制多种DNA重排的潜力。

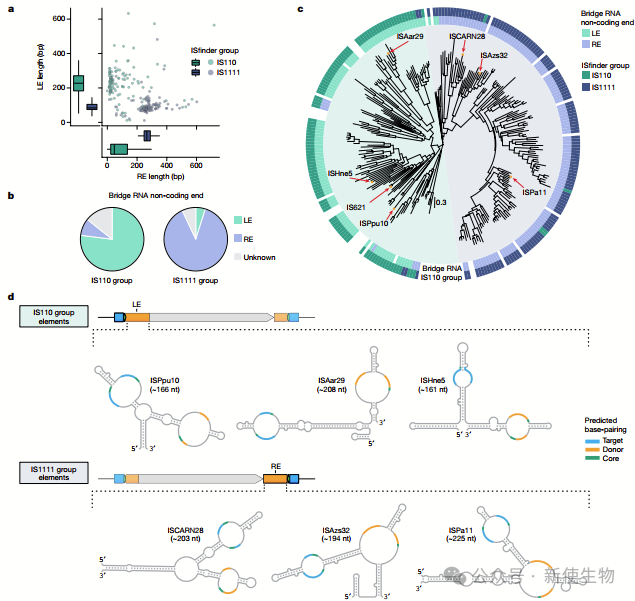
八. 多样的IS110元件编码桥RNA
作者研究了IS110家族中桥RNA的普遍存在。这个家族分为IS110和IS1111两组,后者具有较长的重复元素(RE),与IS110相反。
通过RNA结构模型预测,他们发现大多数IS110和IS1111元素可能编码桥RNA,它们能够特异地与靶位点和供体序列进行碱基配对。
总结
IS110插入序列是一类简化且自主的移动基因元件,通过表达结构化的非编码桥接RNA与其编码的重组酶特异性结合。桥RNA含有两个内部环,能够独立重新编程目标结合环和供体结合环,实现DNA分子之间的特异性序列重组。这一系统不仅支持DNA插入到基因组位点,还能进行可编程的DNA切除和倒置,扩展了核酸引导系统的应用领域,为基因组设计提供了统一的重排机制。
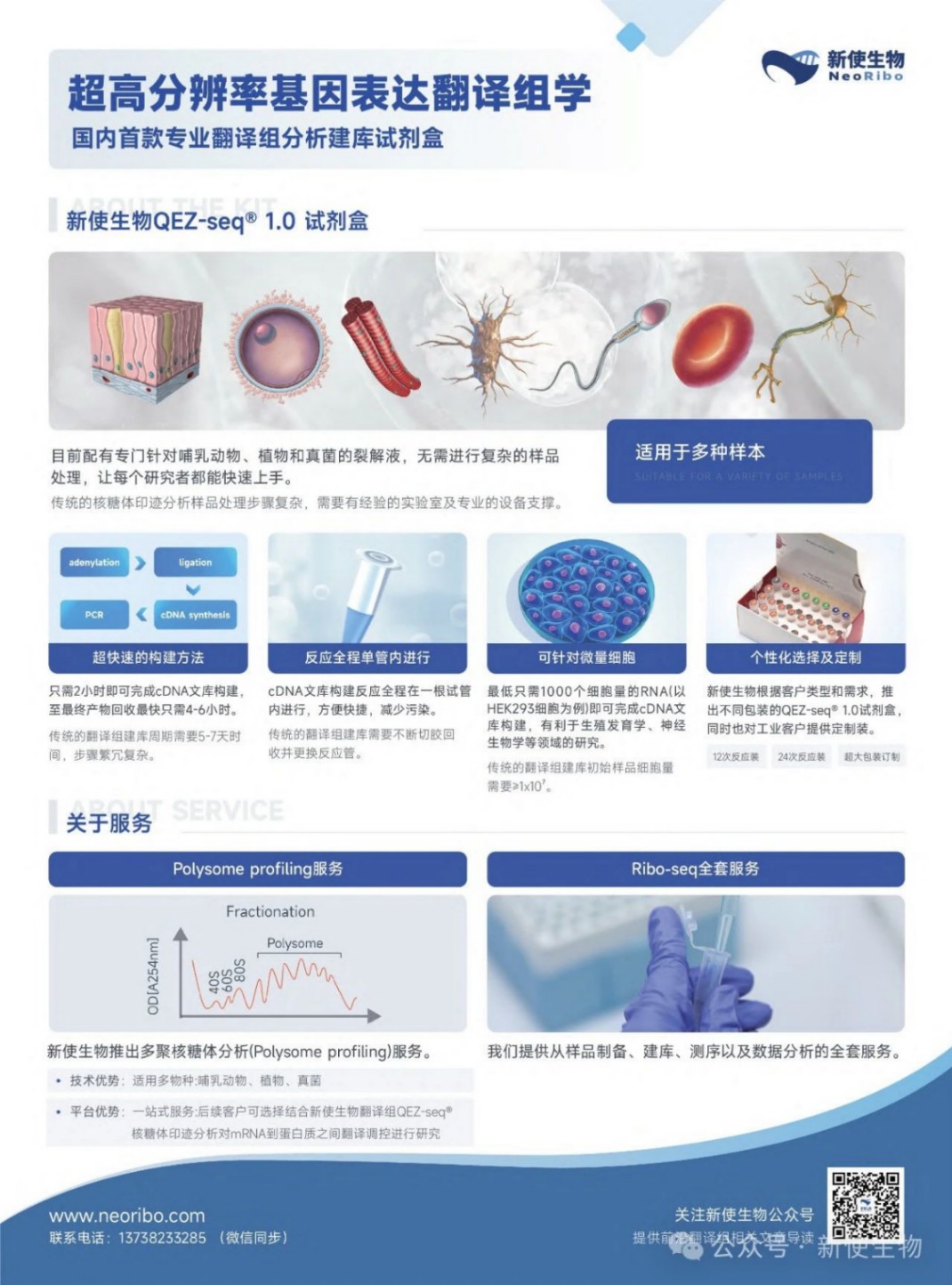
新使生物(neoribo)推出国内首款超高分辨率翻译组ribo-seq建库试剂盒。
我们的核糖体印迹分析技术(QEZ-seq®)适用于哺乳动物、植物、真菌等多类物种,无需复杂操作,建库只需2小时。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
如有咨询翻译组测序请联系杭州新使生物


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们