导读
原核细胞和真核细胞合成的RNA种类繁多,大小和功能各异。RNA分类的一个标准是其编码能力,这区分了编码蛋白质的RNA(mRNA)和非编码RNA(ncRNA)。
非编码RNA包括结构性ncRNA(如rRNA和tRNA)、参与剪接和rRNA生成的snRNA和snoRNA,以及调控性ncRNA(如piRNA、miRNA和lncRNA)。大多数调控性ncRNA小于50个核苷酸,而大于500个核苷酸的称为lncRNA。
RNA-seq可以在转录组水平上检测和定量RNA。文库构建技术大致分为三类:mRNA-seq、Smart-seq和小RNA-seq。
A)在mRNA-seq中,多腺苷酸化(poly(A)+)的mRNA被纯化、片段化,通过随机引物逆转录(RT)转化为cDNA,并连接接头进行扩增和测序。这些方法扩展到非poly(A)转录本,通过去除rRNA并跳过oligo(dT)纯化。
B)Smart-seq基于模板转换,从有限的mRNA中生成高复杂度的文库,包括单细胞的mRNA。
C)mRNA-seq和Smart-seq都无法捕获小RNA,这些小RNA通常通过在5'和3'末端直接连接接头,然后通过RT合成cDNA进行测序。
尽管这种方法已经取得成功,但它只能克隆末端具有特定化学结构的RNA,因为接头连接需要5'端有磷酸基(5'P)和3'端有羟基(3'OH)。
一些ncRNA在末端带有化学修饰,妨碍了标准的连接方法。尽管已经实施了各种策略来克服这一限制,但它们需要事先了解化学末端以进行“修复”。因此,需要一种不依赖连接的小RNA方法,理想情况下还能捕获较长的RNA。
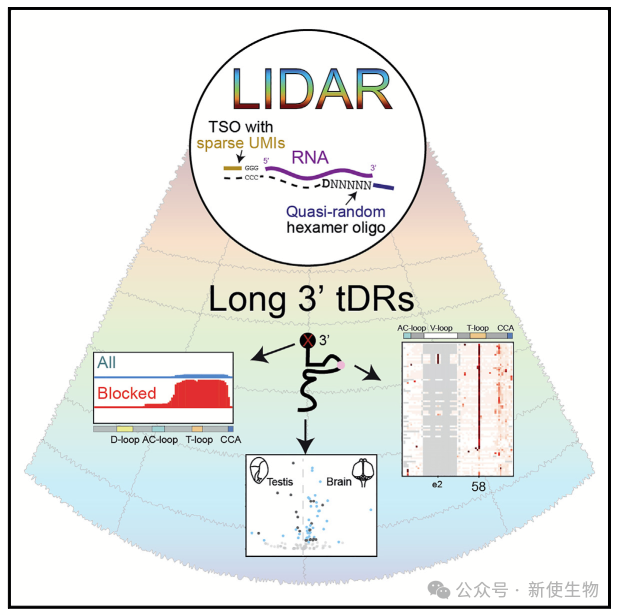
2024年8月2日,宾夕法尼亚大学Roberto Bonasio团队在Molecular Cell上发表了一篇题为“A ligation-independent sequencing method reveals tRNA-derived RNAs with blocked 3′ termini”的论文,介绍了一种新的非连接依赖方法(LIDAR),能检测所有类型的RNA,无论其大小或末端化学结构。LIDAR成功捕获了小鼠胚胎干细胞、组织和精子中的RNA,包括完整的tRNA和之前未检测到的3'末端封闭的tRNA衍生RNA。

文章索引
【标题】A ligation-independent sequencing method reveals tRNA-derived RNAs with blocked 3′ termini
【发表期刊】Molecular Cell
【发表日期】2024年8月2日
【作者及团队】宾夕法尼亚大学Roberto Bonasio团队
【IF】15.58
研究结果
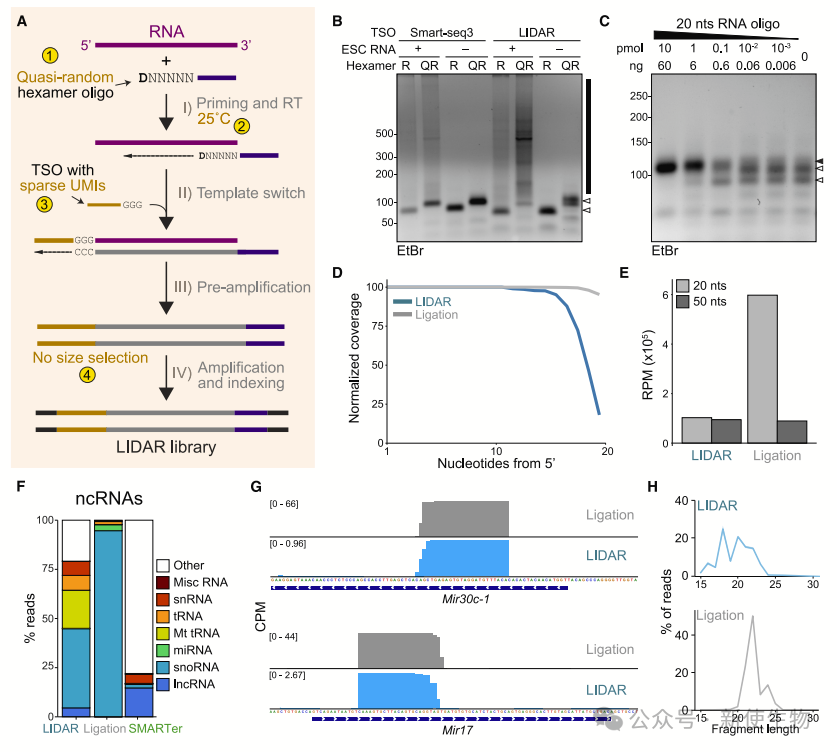
一. LIDAR捕获小RNA
随机六聚体引物广泛用于cDNA合成,但由于难以分离小插入片段与接头二聚体,因此未用于小RNA克隆。LIDAR方法无需尺寸选择即可抑制接头二聚体,从而克隆小RNA。
作者测试了20nt和50nt的RNA,从0.6 ng的输入RNA中获得正确大小的产物,显示出良好的敏感性和覆盖率。
在小鼠胚胎干细胞的总RNA测试中,LIDAR成功构建了文库,包含大量小插入片段(<100 bp),但少于传统连接文库。
LIDAR捕获了大量结构性RNA(如rRNA)和短RNA(如snoRNA、snRNA、tRNA),并能覆盖整个miRNA长度。
总之,LIDAR是一种高效、非连接依赖的方法,可以快速测序各种大小和类型的RNA。
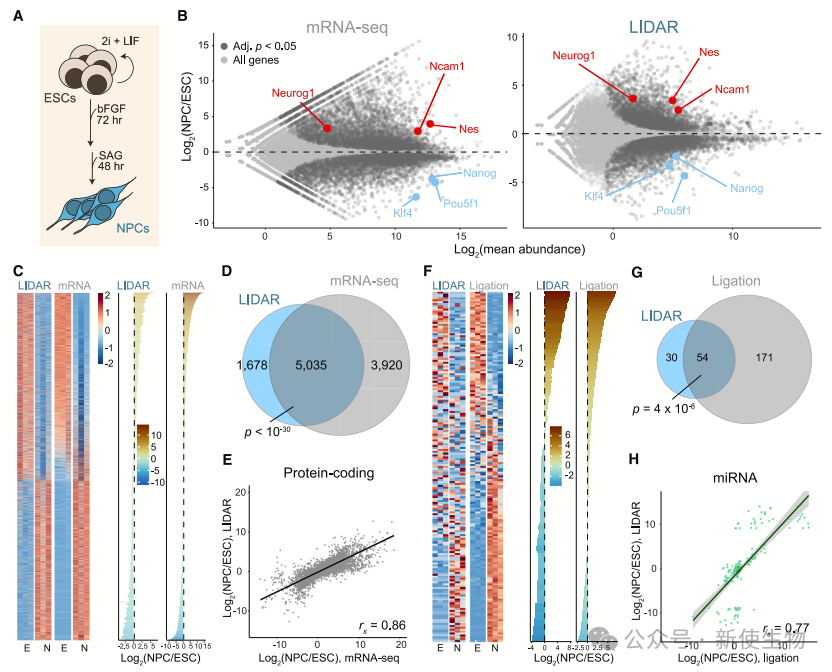
二. 同时检测差异表达的蛋白质编码RNA和小RNA
为了捕获所有大小的RNA,包括小RNA,作者省略了Smart-seq3的标签化步骤,使LIDAR能够检测各种大小的RNA。LIDAR文库排除rRNA后,大部分读取来自mRNA,这在基于连接的方法中几乎无法检测到。
作者将胚胎干细胞(ESCs)分化为神经祖细胞(NPCs),这一过程中基因表达变化显著。
使用LIDAR和传统mRNA-seq,检测到相似数量的差异表达基因,包括多能性基因和NPC标志物。LIDAR与mRNA-seq的表达模式、重叠度和折叠变化相关性都很高,LIDAR还准确量化了miRNA和snoRNA。
总之,LIDAR有效地同时分析了不同RNA群体的基因表达变化,但对于专门分析编码转录组或miRNA,专用方法仍然更好。
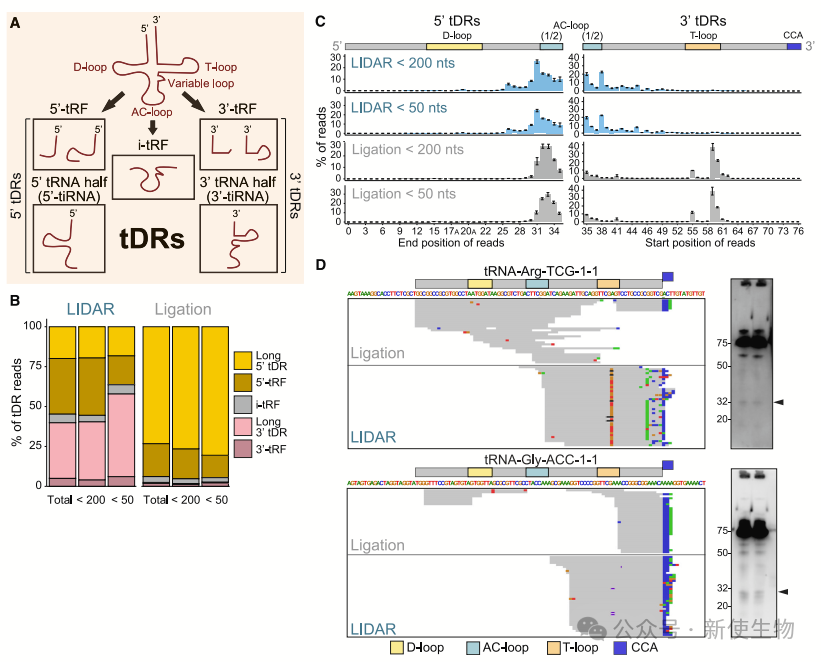
三. LIDAR在胚胎干细胞中揭示了长的3' tDRs
tDRs(tRNA衍生RNA)包括不同类型的3' tDRs。传统的连接方法对3' tDRs检测效率低,LIDAR则能有效捕获这些“无法克隆”的长3' tDRs。
通过LIDAR测序,作者发现了30-40 nt的长3' tDRs,这些片段在传统方法中难以检测。
改进的3'-LIDAR提高了对3'末端的覆盖,能更好地捕获长3' tDRs。LIDAR和3'-LIDAR的结果表明,这些长3' tDRs是在细胞内由成熟tRNA切割产生的。
四. 长3' tDRs在小鼠组织中呈现差异表达
作者研究了长3' tDRs在小鼠脑和睾丸中的表达是否具有组织特异性。
他们使用LIDAR和基于连接的方法构建的文库显示,LIDAR能够检测到更多种类的ncRNA,并且在脑和睾丸中识别出30-40 nt的长3' tDRs。这些长3' tDRs在不同组织中存在差异表达,LIDAR能够有效地量化这些差异。
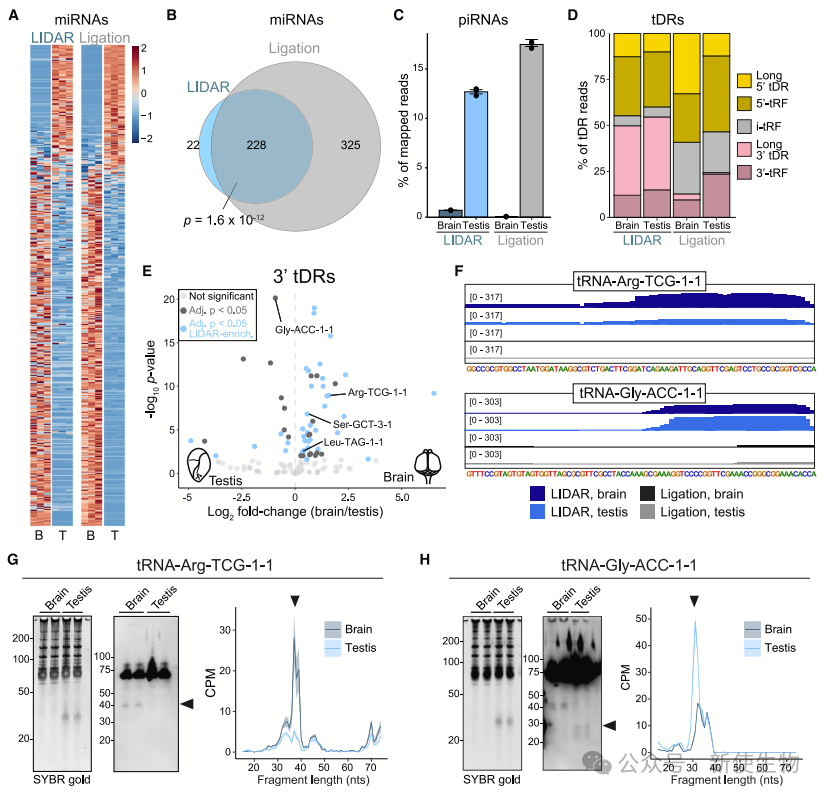
五. LIDAR检测到3' tDRs上的RNA修饰
LIDAR能够检测到3' tDRs上的RNA修饰,如m1A,这些修饰通常会干扰传统的逆转录酶方法。
与PANDORA-seq相比,LIDAR在捕获更多种类的tDRs和检测修饰方面更为出色。LIDAR揭示了30-40 nt的3' tDRs可能来源于成熟tRNA的切割,并且具有修饰。
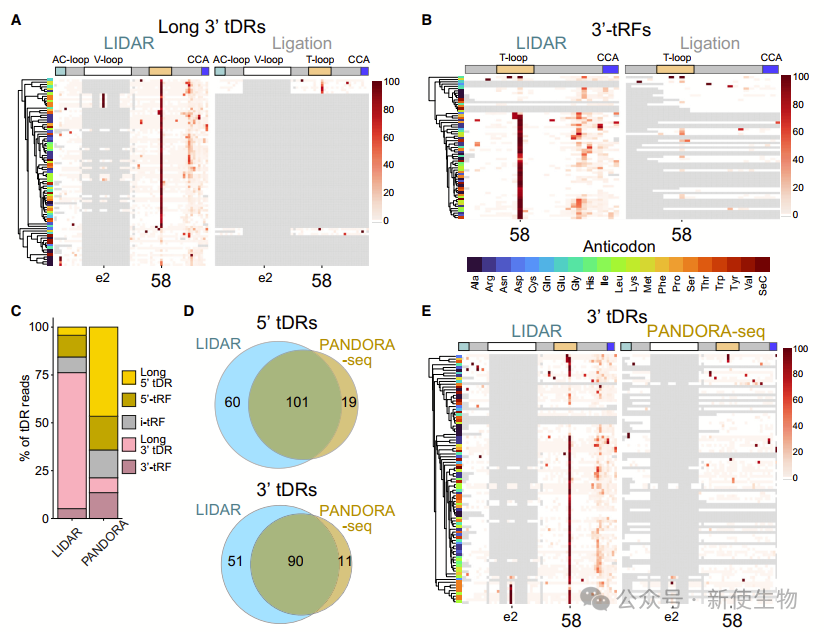
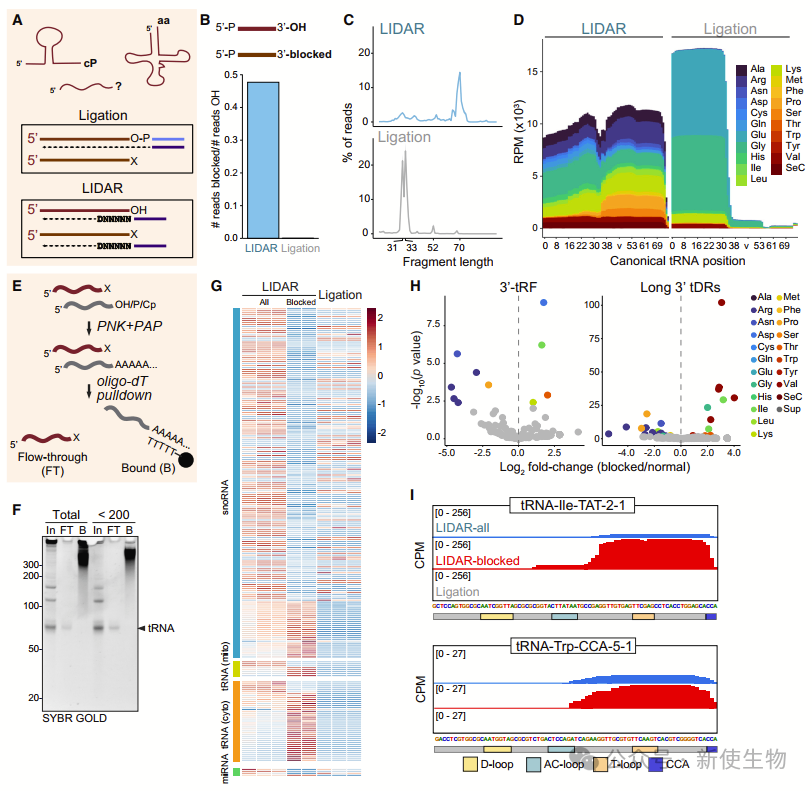
六. LIDAR克隆了3'端封闭的tRNAs和tDRs
LIDAR能够克隆3'端被修饰的RNA,这些修饰使得这些RNA在传统连接方法中难以被检测,而LIDAR有效地捕获了这些3'端封闭的tRNAs和其衍生片段。
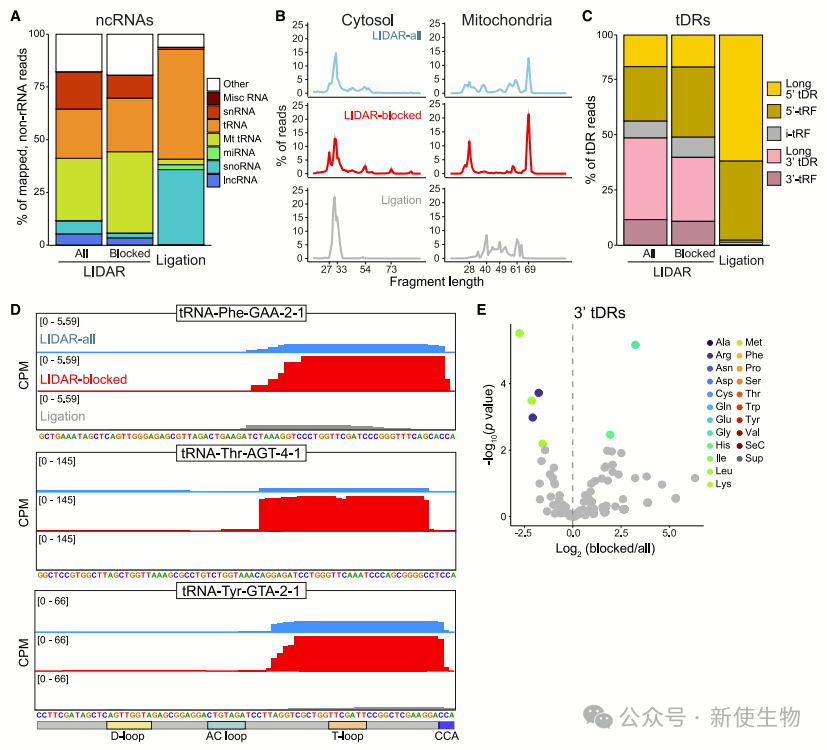
七. LIDAR捕获了小鼠精子的转录本多样性
LIDAR能够捕获小鼠精子中的全面RNA种类,包括传统方法难以检测的3' tDRs和全长tRNAs。
与传统的连接方法相比,LIDAR更有效地识别了被阻断的3'端tDRs,并揭示了精子中的rRNA片段。
LIDAR提供了对精子RNA转录本的更全面分析,发现了在传统和PANDORA-seq方法下未能检测到的长3' tDRs。
总结
本文开发的LIDAR是一种新型测序方法,能够捕获所有长度和30端修饰的RNA,包括编码和非编码RNA。它利用准随机引物扩增和模板转换,克服了传统方法的局限性。LIDAR全面表征了小鼠的转录组,发现了比传统方法更多的tRNA衍生小RNA(tDRs),特别是那些具有阻断3'端的tDRs。这使得LIDAR能够揭示样本中的所有RNA种类及其潜在功能。
新使生物NeoRibo推出国内首款超高分辨率翻译组Ribo-seq建库试剂盒。
我们的Ribo-seq核糖体印迹分析技术(QEZ-seq®)适用于哺乳动物、植物、真菌等多类物种,无需复杂操作,建库只需2小时。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
简便快速的操作让翻译组分析不再设有门槛,每位科研学者都能轻松上手。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可登录 www.neoribo.com



 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们