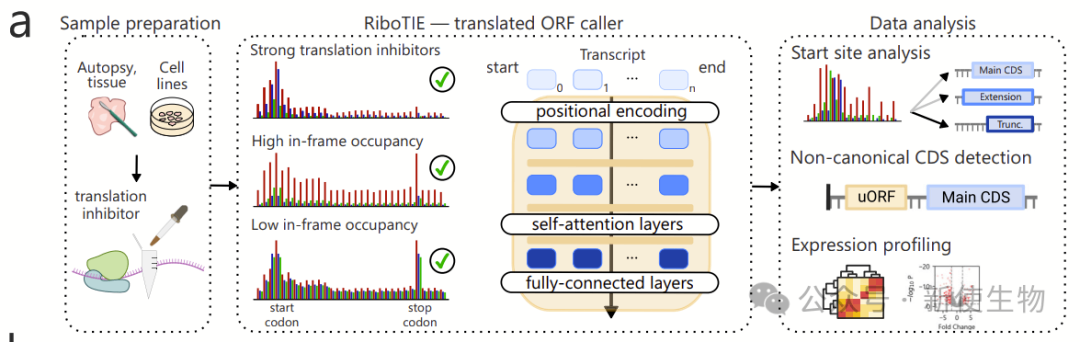
RNA翻译调控决定蛋白质丰度,尤其在癌症等疾病中受多种遗传与非遗传因素影响。RNA翻译是一个复杂的过程,由40S和60S核糖体亚基依次结合RNA,并伴随多种真核起始因子及辅助因子,最终形成活性核糖体。
核糖体印迹分析(Ribo-seq)近年来广泛用于评估mRNA的翻译效率,精准挖掘非典型开放阅读框(ORFs)及可变蛋白形式。因此,对Ribo-seq数据的计算分析已成为多个研究领域的基础,如新基因发现、RNA调控、蛋白质基因组学、微蛋白生物学,以及以RNA翻译为作用靶点的疾病治疗研究。
然而,Ribo-seq的数据分析受限于小ORFs的统计能力不足,以及生物学(如组织类型、细胞系与组织样本的差异)和技术因素(如翻译抑制剂、建库方案)导致的测序读段与翻译模式变异,影响现有工具(如ORFquant、RpBp、Ribo-TISH等)的稳定性,并导致预测分歧和假阳性较多。
2025年2月2日,美国密歇根州大学John R. Prensner团队在Nature Communications上发表了一篇题为“Deep learning to decode sites of RNA translation in normal and cancerous tissues”的论文,开发了RiboTIE工具,改进了基于Ribo-seq核糖体印迹数据ORF的分析,帮助研究者深入挖掘Ribo-seq数据,以拓展对人类细胞及疾病状态的理解。

文章索引
【发表日期】2025年2月2日
【IF】14.7
研究结果
一. RiboTIE优势明显
RiboTIE主要特点:
1)省略预处理步骤,避免数据丢失,提高性能。
2)先预测TIS再构建ORF,确保全转录组覆盖。
3)仅基于RPFs计数分析,减少偏倚。
4)采用Transformer模型与预训练优化性能。
测试显示,RiboTIE在ORF预测的灵敏度和准确性优于现有工具(如ORFquant、RpBp等),识别标准CDS数量提升64.9%,小于300 bp的CDS检出率提高300%。
RiboTIE预测更稳定,假阳性较少,并在u(o)ORFs识别上具有优势,同时减少intORFs和d(o)ORFs的误判。lncRNA-ORFs预测更精准,避免低可信度结果。
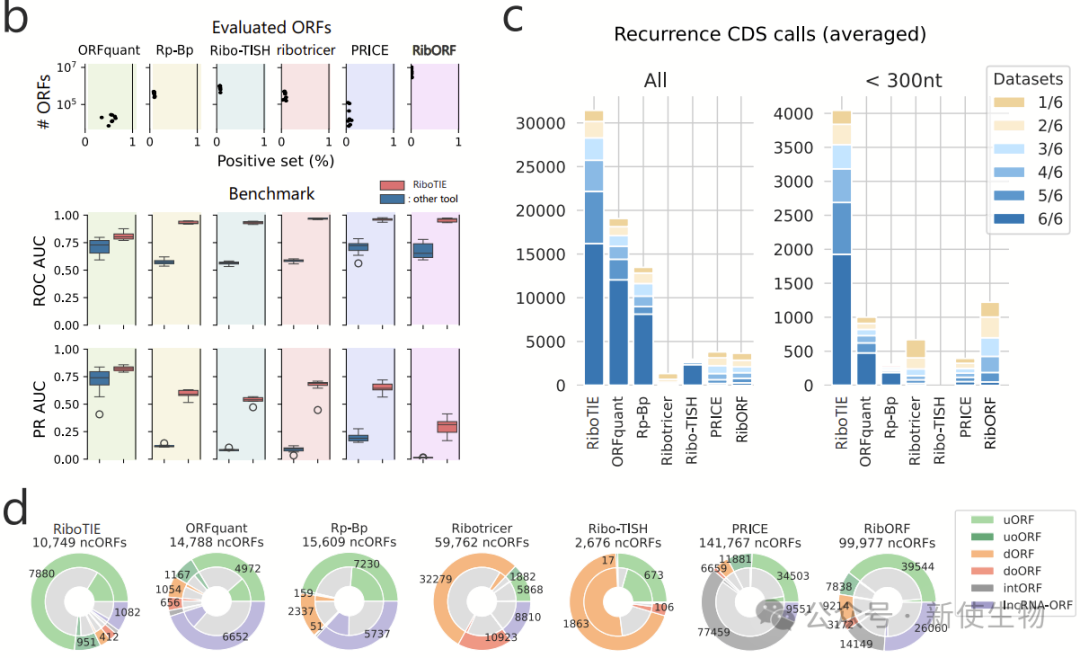
二. 实测能力分析
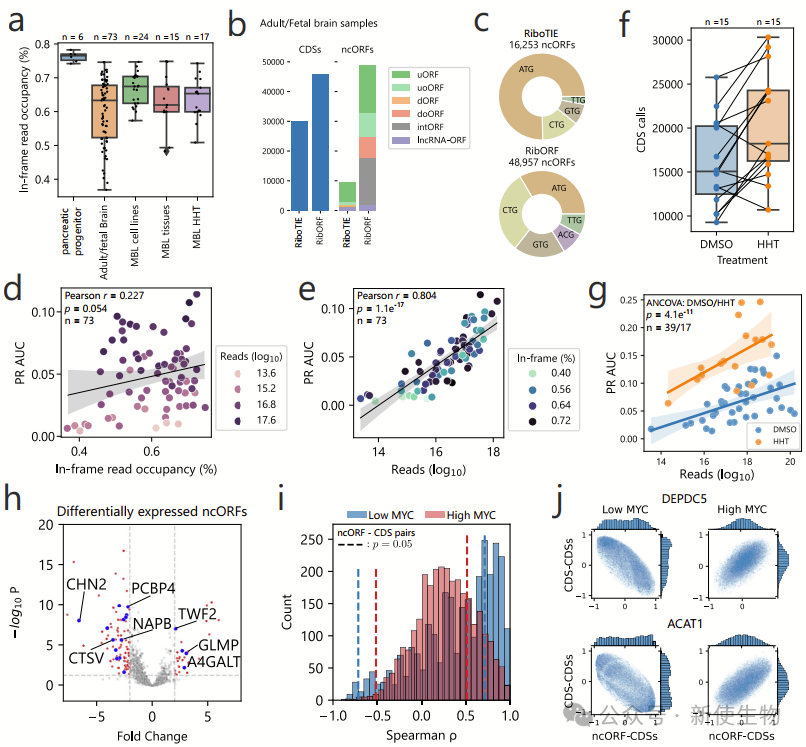
RiboTIE在73例脑组织和15例髓母细胞瘤样本中检测到63786个ORF,相比RibORF预测的158855个ORF,准确性更高。
RiboTIE更依赖TIS周围读段,HHT处理可提高检测性能。分析24例髓母细胞瘤细胞系,鉴定201个MYC相关差异翻译ncORFs,并富集于已知疾病相关基因。
结合TIS Transformer预测22个亚型特异ncORFs,部分ncORF-CDS关联在MYC高/低表达状态下呈反向相关。质谱验证44个ncORF相关多肽,表明ncORFs在髓母细胞瘤中具有调控作用。
总结
RiboTIE是一种基于Transformer模型的方法,可直接利用Ribo-seq核糖体印迹分析测序数据精准检测翻译ORFs,提升分析精度与灵敏度。其应用于正常脑组织和髓母细胞瘤样本,既验证了已知发现,也揭示了新的翻译调控机制,表明RiboTIE可有效解析Ribo-seq数据,助力疾病相关蛋白合成研究。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
简便快速的操作让翻译组分析不再设有门槛,每位科研学者都能轻松上手。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可登录 www.neoribo.com
点击图片查看
点击图片查看


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们