RNA编辑可暂时引入不可遗传的修饰,适用于治疗遗传病和复杂疾病。相较DNA编辑,RNA编辑具有可逆性,避免永久基因组改变,降低脱靶风险,但在面对长期治疗的情况下,RNA编辑的稳定性和效率上仍面临挑战。
目前定点RNA碱基编辑主要有两种策略:A)一是共表达编辑酶与向导RNA(gRNA);B)二是仅递送gRNA以招募内源ADAR,这虽减少脱靶和潜在致癌性,但编辑效率较低。
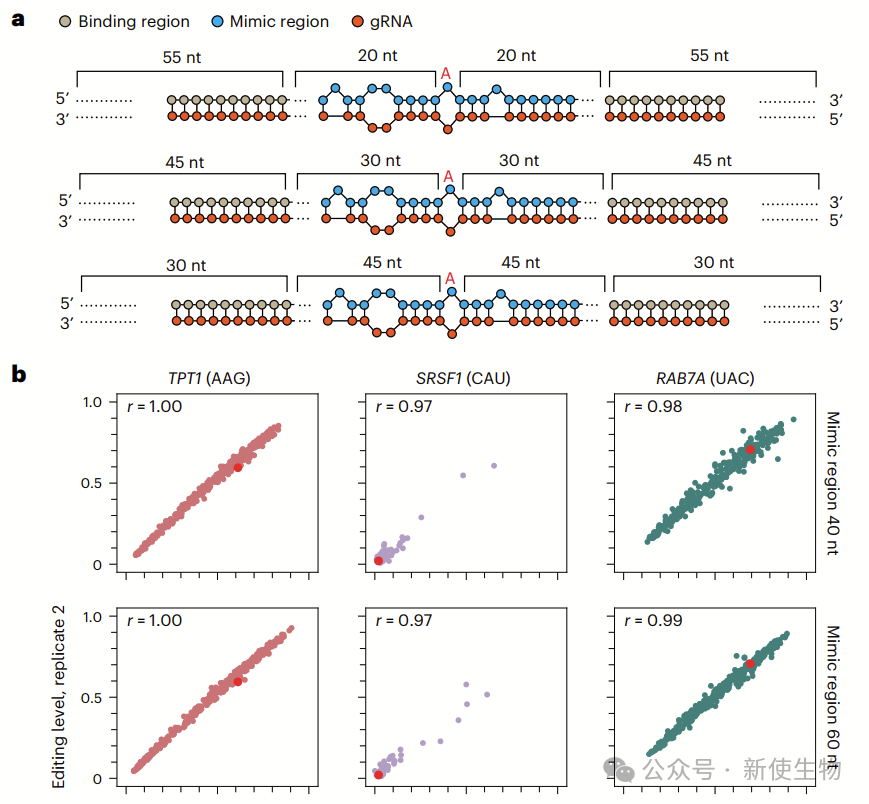
为提高ADAR介导的编辑效率,gRNA设计通常包含一个与靶位点完全互补的区域,并设置A–C错配,有些还引入结构序列以增强ADAR结合。然而,ADAR更依赖RNA结构而非序列,靶位点周围的变异可显著影响编辑效果,因此在gRNA中引入多样结构有助于提高效率,尤其对单gRNA策略至关重要。
1)尽管已有如“−35位点规则”和“−26位点规则”等经验性设计原则,但效率提升有限且靶点依赖性强。
2)直接优化gRNA的策略,如错配扫描,亦受限于结构种类有限及组合空间庞大(如30-mer变异数达10¹⁸),难以通过细胞系统全面筛选。
3)目前主要依赖体外实验,但未能充分模拟ADAR真实环境,因此gRNA设计仍面临显著瓶颈。例如,随机突变法优化的gRNA在细胞中的编辑效率提升也仅约1.5倍。

文章索引
【IF】46.9
研究结果
研究者通过分析GTEx中的RNA-seq数据,识别出30多万对反向Alu序列,富集高度RNA编辑位点,具备典型ADAR编辑特征。
实验结果验证了这些反向Alu是理想的内源编辑模板,可用于指导高效gRNA设计。
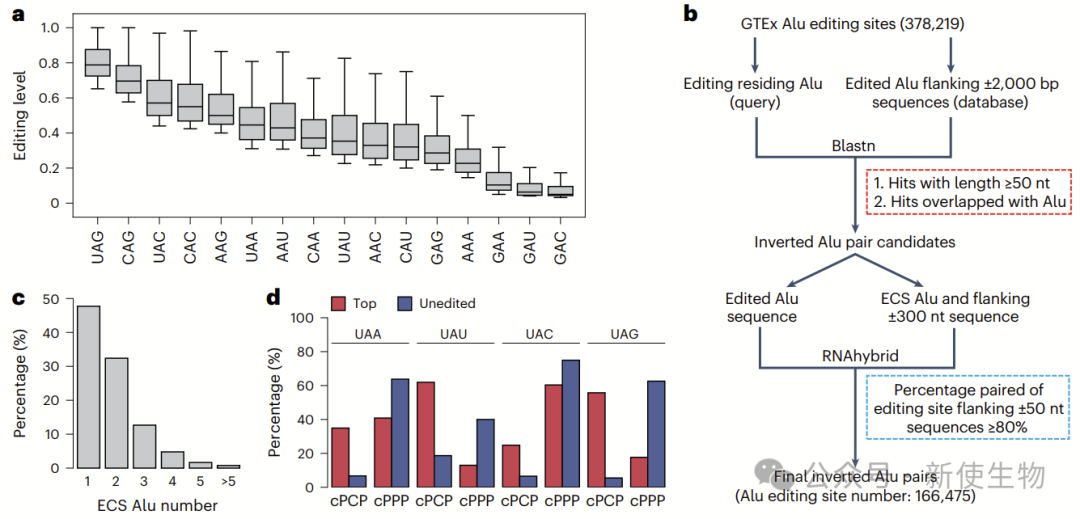
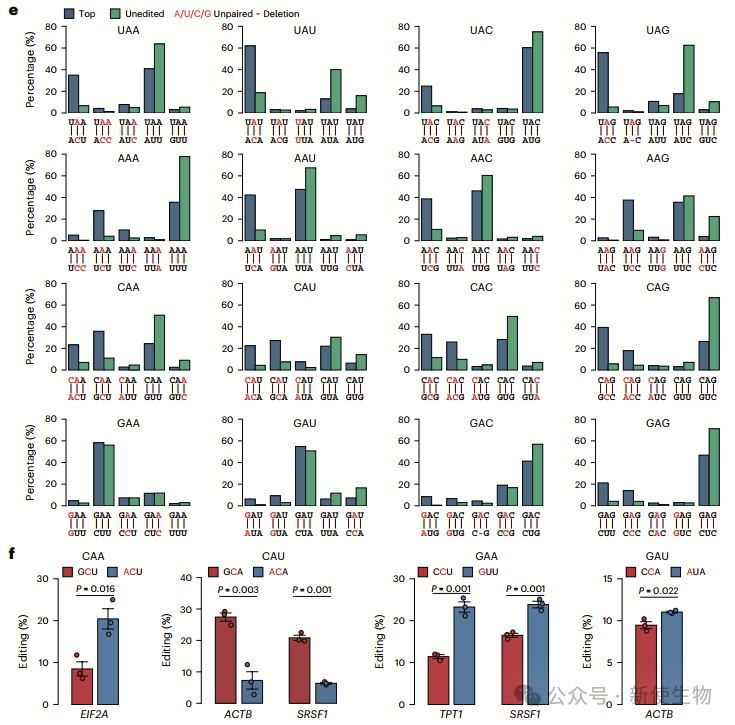
作者发现,高编辑位点具有特定的三联序列特征,为gRNA设计提供了新线索。大多数位点富集cPCP序列,但对于GAN位点,替代设计(如−1位错配G–G或G–A)表现更好。
实验验证表明,某些替代设计在天然底物中比cPCP设计具有更高的编辑效率,提供了优化gRNA设计的潜力。
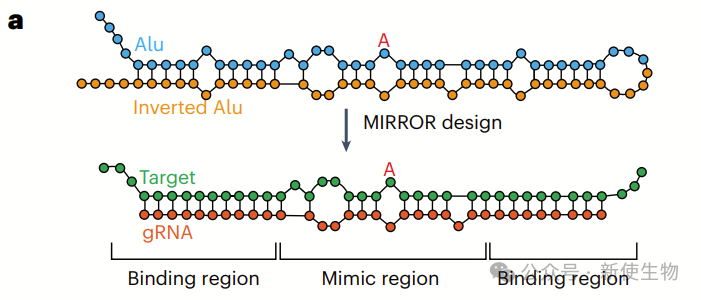
作者提出了MIRROR方法,通过设计gRNA模拟倒位重复序列的结构,优化RNA编辑效率。
该方法通过构建一个高通量筛选系统,评估gRNA在特定底物上的编辑效率,验证了结构特征对编辑效率的贡献。
实验结果表明,gRNA的编辑效率与底物的结构和未配对碱基的数量密切相关,最佳的结构设计可显著提高编辑效果。
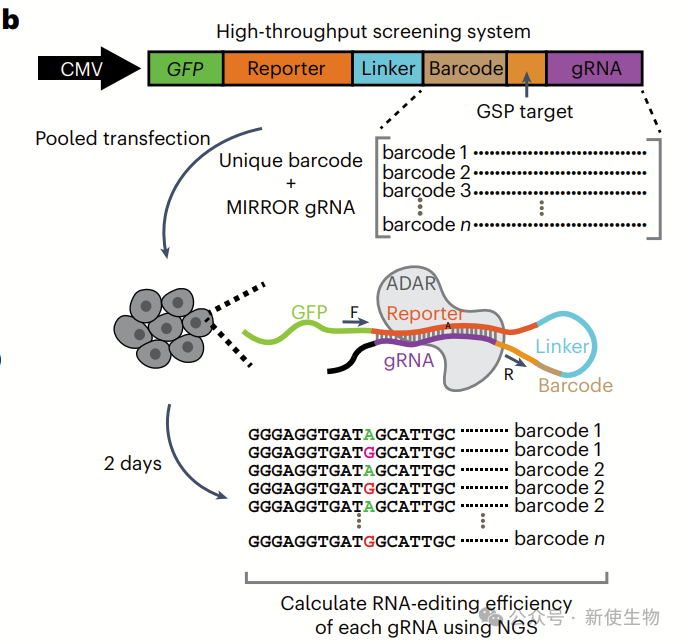
为了优化短gRNA的RNA编辑效率,作者采用了MIRROR策略,并对41 nt长的gRNA进行了筛选,重点研究了不同位置的编辑效应。
他们首先在HEK293细胞中测试了这些gRNA,发现不同细胞系中gRNA的编辑效率高度一致,表明RNA编辑的结构规则在多种细胞类型中是保守的。
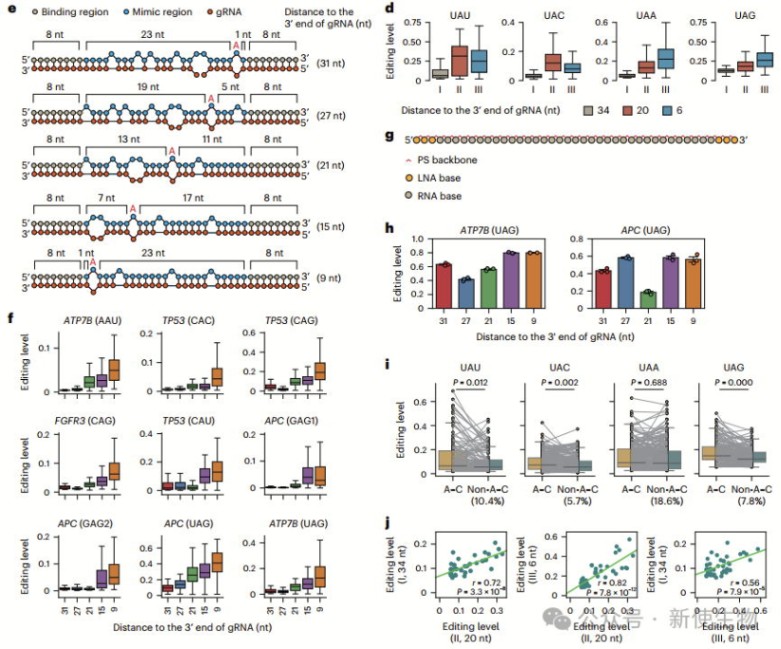
接着,他们发现编辑效率受gRNA 3′区域偏好影响,gRNA的3′端与目标位点的编辑效果最佳。
进一步使用化学修饰的gRNA测试后,团队确认了gRNA结构对编辑效率的提升作用,尤其是与ADAR结合的最佳结构。
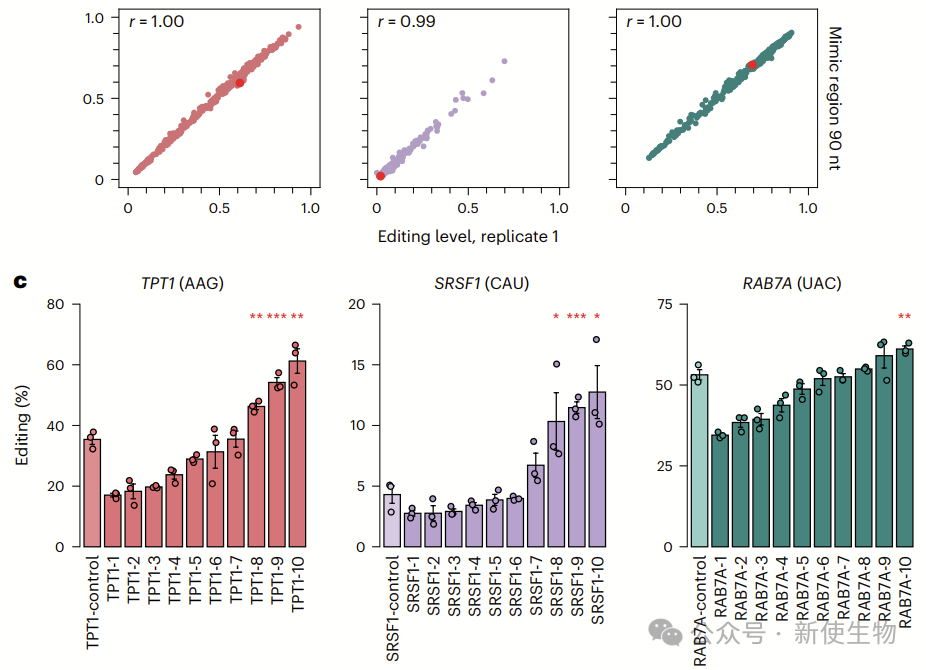
最终,作者验证了不同病变位点的编辑效率,发现最优MIRROR gRNA在多个病变位点上表现出显著的编辑效率提升,比传统设计高2.5到5.7倍。
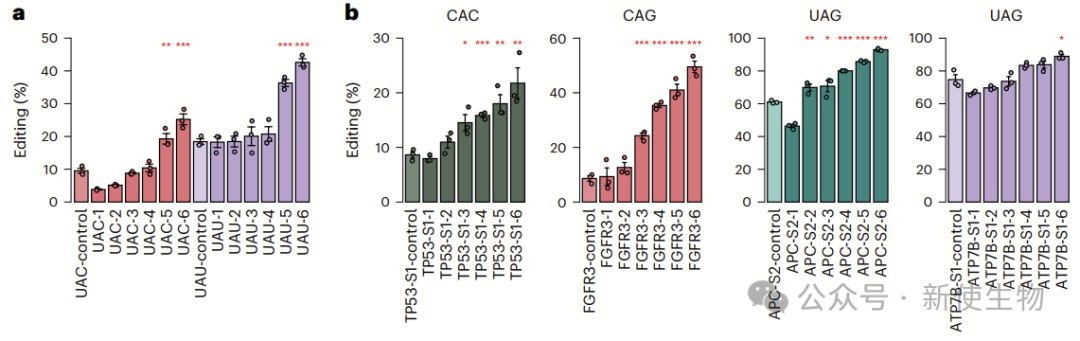
为了验证MIRROR gRNA的编辑能力,作者设计了针对Z等位基因的SERPINA1 mRNA的MIRROR gRNAs。
结果表明,MIRROR gRNAs在不同长度和浓度下均显示出比完全互补的A–A错配gRNAs更高的编辑效率,最高提高了2倍。
MIRROR gRNA通过脂质纳米颗粒递送,在小鼠体内实现高达81%的编辑效率,且编辑效果在7天内保持稳定,这表明MIRROR gRNA能有效在体内进行RNA编辑。

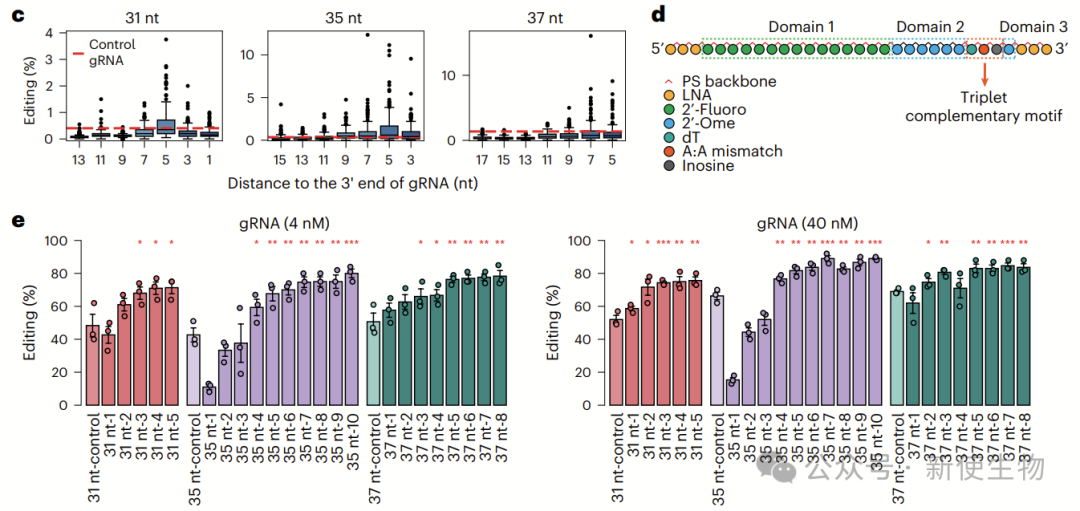
团队验证了MIRROR策略在质粒系统生成长gRNAs中的应用,结果表明,MIRROR gRNAs显著提高了编辑效率,尤其是针对非TAN模体的gRNAs,效率分别提高了1.7倍和2.8倍。
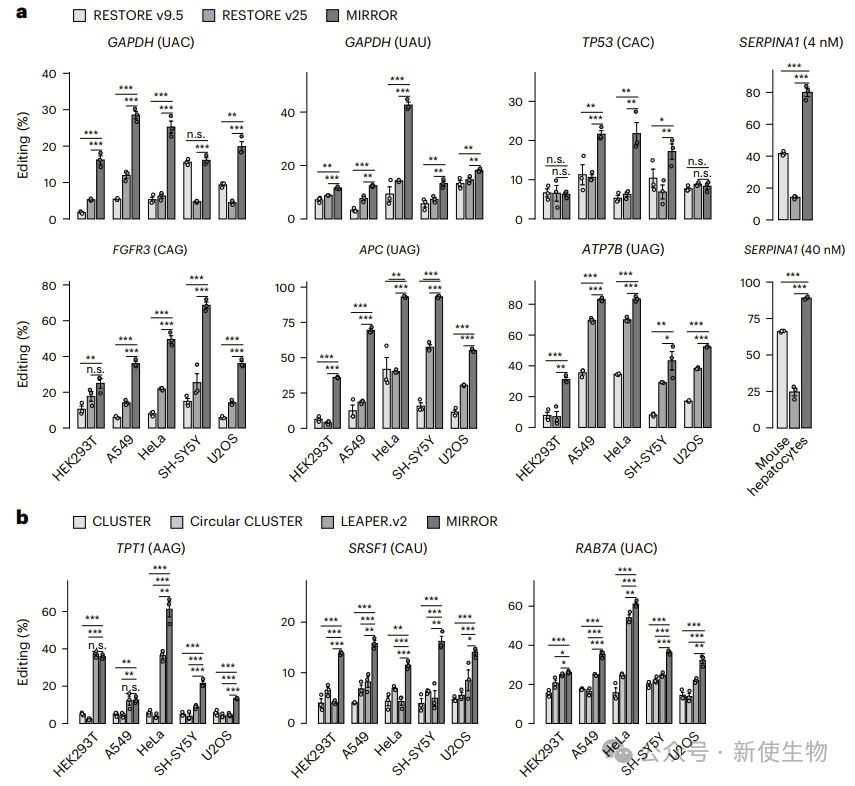
作者对MIRROR在多种细胞类型中与现有设计方法进行了比较,结果显示,在短化学合成gRNA和长生物生成gRNA形式下,MIRROR的编辑效率普遍高于其他方法。
MIRROR在大多数情况下提高了编辑效率,超越了RESTORE、LEAPER.v2、CLUSTER和圆形CLUSTER设计,证明其是一种适用于广泛细胞类型和生物体的高效工具。
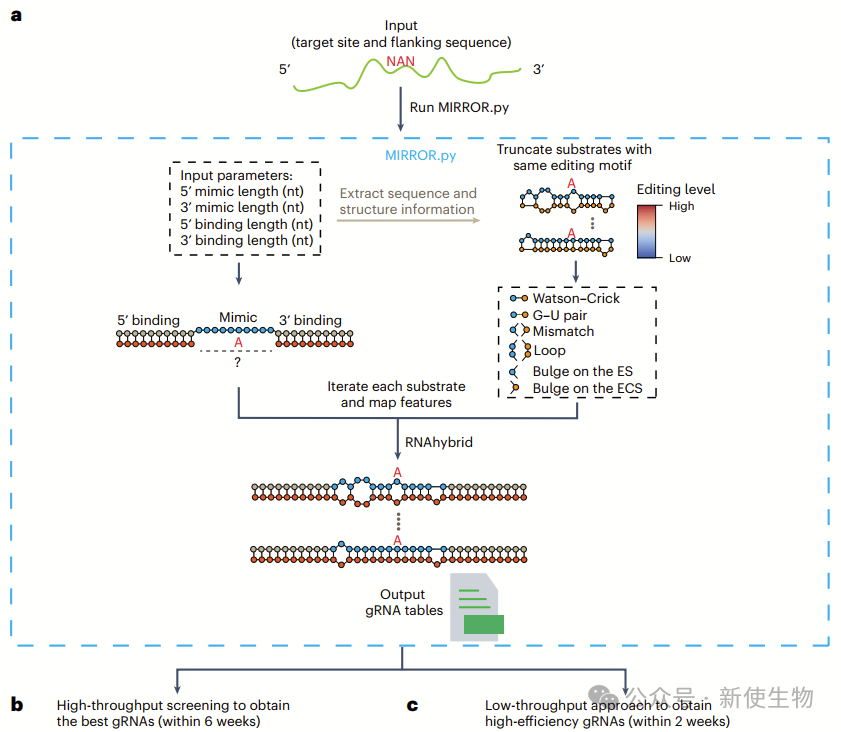
为了简化MIRROR gRNA的设计,作者开发了一个程序,用户输入目标编辑位点和序列,程序自动生成1000-2000个候选gRNA。高通量筛选方法需要大约6周,低通量筛选方法则大约2周即可获得最佳gRNA。
作者还通过训练XGBoost模型,结合SHAP值分析,识别出提升编辑效率的关键特征,这些特征可应用于低通量筛选的gRNA设计。
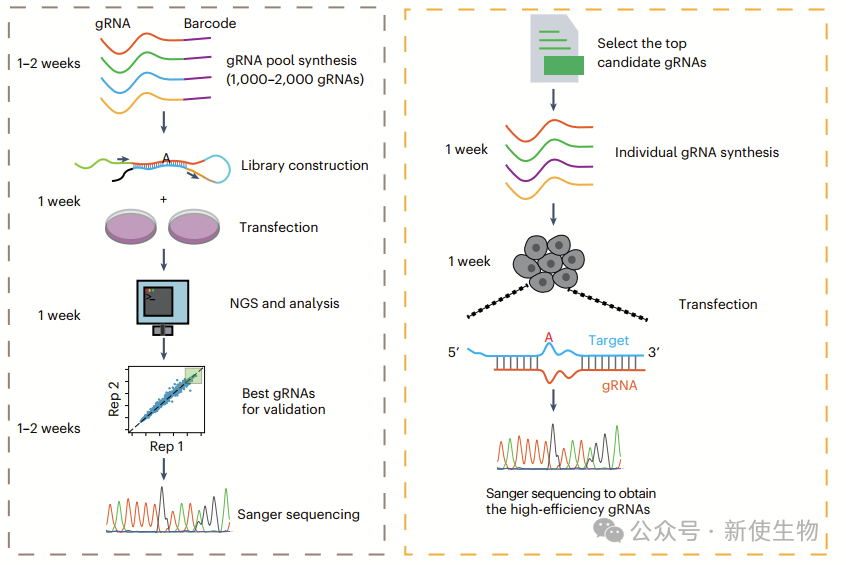
本文介绍了MIRROR方法,通过模仿倒位的Alu重复序列结构,优化了gRNA设计以招募ADAR进行RNA碱基编辑。与现有方法相比,MIRROR显著提高了RNA编辑效率,适用于短链化学合成和长链生物合成gRNA,在多种人类细胞和小鼠肝细胞中表现出色。
我们能够针对微量细胞或组织,如卵母细胞、卵巢、临床穿刺样品等产出高质量翻译组数据结果。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可登录 www.neoribo.com
点击图片查看
点击图片查看


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们